El asma es una afección inflamatoria de las vías respiratorias. Las infecciones por Mycoplasma pneumoniae pueden exacerbar los síntomas del asma. Se ha demostrado que la interleucina2 y la interleucina4 participan en las reacciones inmunitarias e inflamatorias. Hemos estudiado la relación entre los polimorfismos de la IL2 y la IL4 y su expresión y el riesgo de padecer asma e infección por M.pneumoniae en niños.
MétodosSe reclutó a 392 niños asmáticos y 849 controles para el estudio. Se genotiparon 8 polimorfismos en IL2 e IL4 con la plataforma MassARRAY de Sequenom. La infección por M.pneumoniae y el número de copias se establecieron mediante PCR fluorescente. Los niveles séricos de expresión de IL-2 e IL-4 se midieron con ELISA.
ResultadosHallamos una relación significativa entre el polimorfismo rs6534349 de IL2 y el aumento de riesgo de sufrir asma (heterocigóticos, p=0,029; variantes homocigóticas, p=0,013), así como entre el polimorfismo rs2227284 de IL4 y una reducción del riesgo de padecer asma (heterocigóticos, p=0,026; variantes homocigóticas, p=0,001). Además, la relación con otros polimorfismos, excepto el rs2070874, se hizo evidente al agrupar a los niños asmáticos según la clasificación GINA de control y gravedad del asma. Asimismo, los niveles séricos de expresión de IL-2 e IL-4 fueron significativamente mayores en los sujetos no infectados (p=0,038) e infectados (p=0,011) por M.pneumoniae, respectivamente. Esta observación también se cumple entre los pacientes asmáticos (p=0,016 para IL-2 y p=0,042 para IL-4), pero en los controles no asmáticos solo se cumple en el caso de la IL-4 (p=0,032). Del mismo modo, observamos que el genotipo GG rs6534349 estaba claramente relacionado con un aumento de las posibilidades de tener una infección con alta carga de M.pneumoniae (p=0,0376).
ConclusionesLa IL2 y la IL4 podrían ser biomarcadores importantes para calcular el riesgo de padecer asma, así como infección por M.pneumoniae, en niños.
Asthma is an inflammatory disorder of the airways and the symptoms of asthma could be exacerbated by Mycoplasma pneumoniae infection. Interleukin-2 and interleukin-4 have been implicated in immune and inflammatory reactions. We examined the associations of IL2 and IL4 polymorphisms and expression with the risks of asthma and M. pneumoniae infection in children.
Methods392 asthmatic children and 849 controls were recruited into the study. Eight polymorphisms in IL2 and IL4 were genotyped with Sequenom MassARRAY platform. M. pneumoniae infection and copy number was determined with fluorescence PCR. IL-2 and IL-4 serum expression levels were determined by using ELISA.
ResultsWe found a significant association of IL2 rs6534349 polymorphism with increased asthma risk (heterozygotes, P=.029; homozygous variants; P=.013) and of IL4 rs2227284 polymorphism with reduced asthma risk (heterozygotes, P=.026; homozygous variants; P=.001). Besides, the association of other polymorphisms, except rs2070874 polymorphism, became apparent when the asthmatic children were grouped according to GINA classification of asthma control and severity. In addition, IL-2 and IL-4 serum expression levels were significantly higher in M. pneumoniae negative (P=.038) and positive (P=.011) subjects respectively. This observation holds true among asthmatic patients (P=.016 for IL-2 and P=.042 for IL-4), but only the IL-4 observation remained correct among non-asthmatic controls (P=.032). We also observed that the rs6534349 GG genotype was significantly associated with increased odds of getting high load M. pneumoniae infection (P=.0376).
ConclusionsIL2 and IL4 could be important biomarkers for estimating the risks of asthma and M. pneumoniae infection in children.
El asma es una afección inflamatoria crónica compleja de las vías respiratorias caracterizada por obstrucción respiratoria e hiperreactividad. En los niños, el asma suele aparecer por inflamaciones alérgicas y puede causar distintos grados de obstrucción respiratoria, como disnea, tos, tensión torácica y episodios recurrentes de sibilancia, entre otros. En China, al igual que en el resto del mundo, el asma infantil es una preocupación importante de salud pública dentro de la neumología pediátrica. La prevalencia del asma infantil ha aumentado anualmente1,2.
Mycoplasma pneumoniae es un patógeno importante, conocido principalmente como agente causante de la neumonía extrahospitalaria, en especial entre los niños3. La sospecha de la relación entre la infección por M.pneumoniae y el asma existe desde hace décadas4. Más recientemente han surgido varias líneas de evidencia que implican a la infección por M.pneumoniae en la aparición y la exacerbación del asma5,6. La infección por M.pneumoniae causa lesiones en las células epiteliales respiratorias, lo que aumenta la reactividad de las vías respiratorias y activa un gran número de linfocitosT7. Los linfocitosT fomentan la activación de los linfocitosB que producen anticuerpos y liberan citoquinas inflamatorias, lo que, a su vez, contribuye a la aparición de síntomas asmáticos.
A pesar de la complejidad de los mecanismos subyacentes a la manifestación del asma, se sabe que las interleucinas (IL), en especial IL-2 e IL-4, desempeñan un papel central en este proceso8. La IL-2 favorece la maduración de los linfocitos T primitivos, mientras que la IL-4, mediante su compleja interacción con la IL-12, ayuda a tomar la importante decisión de si los linfocitosT deben diferenciarse en Th1 o Th2 que producen distintos tipos de citoquinas9. Estos eventos causan cambios fisiopatológicos importantes durante el asma y, por lo tanto, son un paso clave en su evolución. Además, el papel destacado de la IL-2 y la IL-4 en el sistema inmunitario adaptativo sugiere que las citoquinas podrían influir en la eficiencia de las células inmunitarias a la hora de rechazar las infecciones por M.pneumoniae. Por lo tanto, planteamos la hipótesis de que la variación del nivel de IL-2 e IL-4 y su actividad podría relacionarse con el riesgo de padecer asma e infección por M.pneumoniae10,11. Un factor potencial que podría influir en la variación interindividual del nivel y actividad de IL-2 e IL-4, y por lo tanto en el riesgo de padecer asma, son los polimorfismos de nucleótido simple (SNP por sus siglas en inglés) dentro de los genes que codifican las citoquinas12,13. En este estudio intentamos establecer la relación entre los SNP en los genes de IL2 e IL4 y el riesgo de padecer asma en niños. En concreto, nos planteamos estudiar los polimorfismos rs6822844, rs6534349, rs2069762 y rs3136534 de IL2 y rs2243250, rs2070874, rs2227284 y rs2243290 de IL4. También deseamos investigar la presencia de niveles séricos diferenciales de expresión de IL-2 e IL-4 en pacientes infectados y no infectados por M.pneumoniae y estudiar la relación entre los mismos SNP y el riesgo de infecciones con alta carga de M.pneumoniae.
MétodosSujetosEl estudio contó con la aprobación del Comité Ético del Hospital Materno-infantil Yiwu en Zhejiang (Ref. 2009/PED/0215.045). Se seleccionaron a un total de 392 niños con asma y 849 controles no asmáticos con edades comprendidas entre los 4 y los 15años del Hospital Materno-infantil Yiwu y el Hospital Pediátrico de la provincia de Zhejiang entre marzo de 2009 y octubre de 2013. Se identificó a los niños con asma mediante diagnóstico rutinario basado en las directrices de Iniciativa Global contra el Asma (GINA). Los controles fueron niños sin asma ni síntomas de alergia que habían acudido a estos mismos hospitales por otras cuestiones médicas no relativas al asma. En total, entre los controles, 83 presentaban estridor; 89, aspiración de cuerpo extraño; 80, tos persistente; 66 fueron diagnosticados con infección pulmonar, y 531 eran niños sanos que acudieron al hospital para un seguimiento tras ser diagnosticados con una infección respiratoria transitoria. Se ajustaron los controles y casos por frecuencia en cuanto a género y edad. Todos los sujetos eran chinos de etnia han. Los padres firmaron el consentimiento informado antes de la inclusión de los sujetos en el estudio.
Detección de infección por Mycoplasma pneumoniaeSe extrajo ADN de las muestras respiratorias (extensión faríngea, n=528; lavado broncoalveolar, n=432; muestras de esputo, n=204; aspiración bronquial, n=77) de los sujetos. Se realizó lavado broncoalveolar en 318 controles (es decir, los que presentaban estridor, aspiración de cuerpos extraños, tos persistente o con diagnóstico de infección pulmonar) como parte de su tratamiento terapéutico/diagnóstico, así como en 114 niños asmáticos para identificar los agentes infecciosos y los perfiles celulares y de citoquinas (por motivos diagnósticos o como parte de otra investigación en curso). Se realizó un único lavado broncoalveolar por paciente. Para detectar la infección por M.pneumoniae en los sujetos se utilizó el Fluorescence PCR Mycoplasma pneumoniae Detection Kit (Acon Biotech, Hangzhou, China), siguiendo las instrucciones del fabricante.
Cuantificación de IL-2 e IL-4 por ELISASe aisló el suero de las muestras de sangre recogidas y se diluyó 1:4 en el diluyente de la muestra antes de emplearlo para cuantificar los niveles de IL-2 e IL-4 por ELISA. Las concentraciones séricas de IL-2 e IL-4 se establecieron con los kits ELISA Human Interleukin 2/IL-2 y ELISA Human Interleukin 4/IL-4 (Xinqidi Biological Technology Co. Ltd., Wuhan, China) con una sensibilidad de <0,40pg/ml y <0,20pg/ml, respectivamente, siguiendo los protocolos del fabricante.
Genotipado de los polimorfismos de nucleótido simpleSe aisló el ADN de las muestras de sangre de los sujetos con el kit genético TIANamp (Tiangen Biotech, Beijing, China). Se genotiparon los polimorfismos en el ADN aislado usando la plataforma MassARRAY (Sequenom, San Diego, CA, EE.UU.), siguiendo las instrucciones del fabricante. Se repitió el genotipado en el 10% de las muestras por motivos de confirmación. La tasa de reproducibilidad de los genotipos fue del 100%.
Análisis estadísticoLa distribución de los genotipos en los niños asmáticos y los controles no asmáticos se comparó mediante la prueba de χ2, y la asociación entre los polimorfismos y el riesgo de padecer asma se evaluó mediante análisis de regresión logística. Para estudiar la relación entre los polimorfismos y el riesgo de predisposición a infecciones con alta carga de M.pneumoniae, se comparó la distribución genotípica mediante la prueba de χ2 seguida de análisis de regresión logística. Las expresiones de IL-2 e IL-4 en pacientes infectados por M.pneumoniae y no infectados por M.pneumoniae se compararon mediante la prueba t para determinar si existe diferencia significativa en la expresión entre ambos grupos. En todos los análisis se consideró que los resultados eran significativos cuando p<0,05.
ResultadosCaracterísticas de los sujetos e infección por Mycoplasma pneumoniaeSe ajustaron los controles y casos por frecuencia en cuanto a género y edad. No hubo diferencias significativas entre casos y controles en cuanto a género (p=0,99) y edad promedio (p=0,98). Conforme a la clasificación GINA, 177 casos correspondían a asma controlada; 118, a asma parcialmente controlada, y 97, a asma no controlada. Además, 102 se clasificaron como grave; 80, como moderada, y 210, como leve.
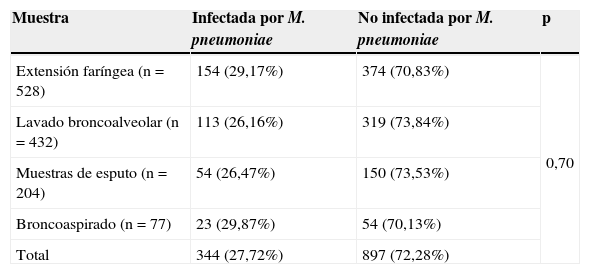
Detección de infección por Mycoplasma pneumoniaeUn total de 344 dieron positivo para infección por M.pneumoniae, de los que 130 eran niños asmáticos y 214, controles no asmáticos. Por otro lado, de los 897 sujetos restantes no infectados por M.pneumoniae, 262 eran niños asmáticos y 635, controles no asmáticos. En la tabla 1 se muestra la prevalencia de M.pneumoniae detectada en los distintos tipos de muestras. La muestra empleada no afectaba de forma significativa a la detección de M.pneumoniae (p=0,70).
Detección de la infección por Mycoplasma pneumoniae empleando distintas muestras
| Muestra | Infectada por M. pneumoniae | No infectada por M. pneumoniae | p |
|---|---|---|---|
| Extensión faríngea (n=528) | 154 (29,17%) | 374 (70,83%) | 0,70 |
| Lavado broncoalveolar (n=432) | 113 (26,16%) | 319 (73,84%) | |
| Muestras de esputo (n=204) | 54 (26,47%) | 150 (73,53%) | |
| Broncoaspirado (n=77) | 23 (29,87%) | 54 (70,13%) | |
| Total | 344 (27,72%) | 897 (72,28%) |
El número de copias bacterianas en los sujetos infectados por M.pneumoniae osciló entre 6,43×103 y 6,48×107 organismos ml−1, con una mediana y un promedio de 3,96×105 y 1,67×107 organismos ml−1, respectivamente; 206 y 138 sujetos tenían una carga bacteriana inferior o superior a la media, respectivamente.
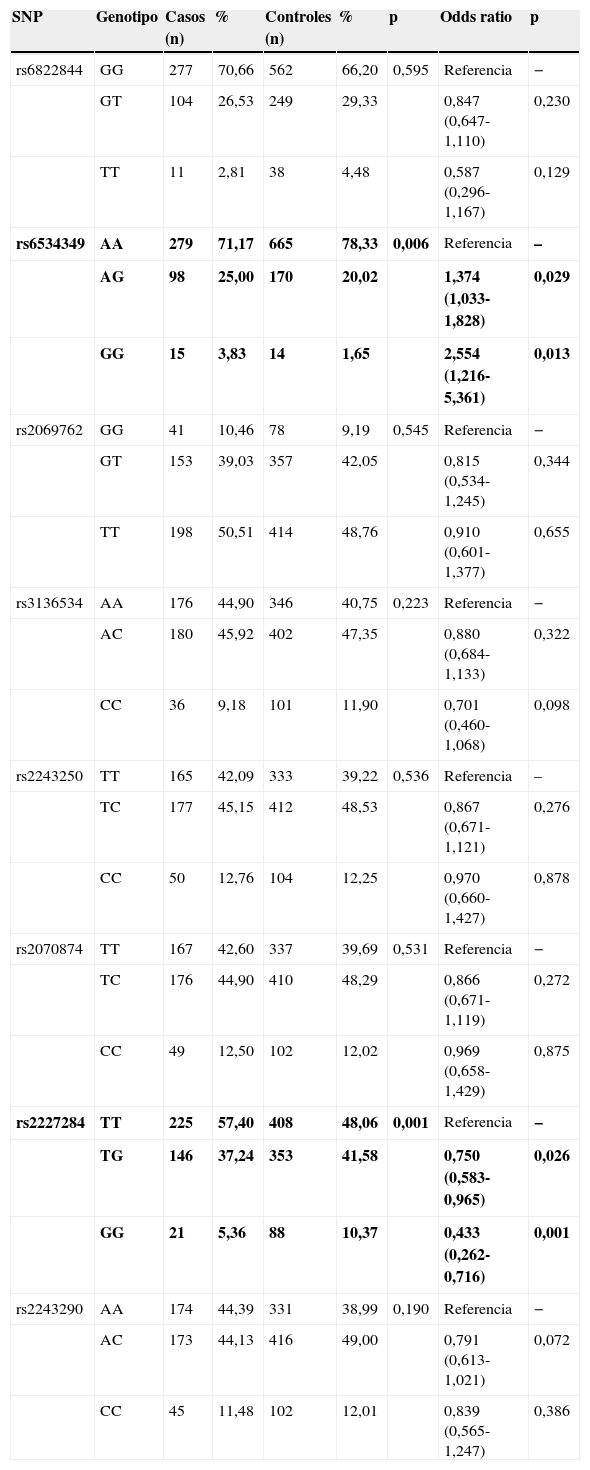
Distribución genotípica en niños asmáticos y controles sanosEntre los 8 polimorfismos estudiados, solo se observó una diferencia significativa entre casos y controles en 2 polimorfismos: el polimorfismo rs6534349 de IL2 (p=0,006) y el polimorfismo rs2227284 de IL4 (p=0,001) (tabla 2). No se encontró desviación del equilibrio de Hardy-Weinberg para ninguno de los 8 polimorfismos (p>0,05).
Distribución genotípica y asociación de riesgo en niños asmáticos y controles sanos
| SNP | Genotipo | Casos (n) | % | Controles (n) | % | p | Odds ratio | p |
|---|---|---|---|---|---|---|---|---|
| rs6822844 | GG | 277 | 70,66 | 562 | 66,20 | 0,595 | Referencia | − |
| GT | 104 | 26,53 | 249 | 29,33 | 0,847 (0,647-1,110) | 0,230 | ||
| TT | 11 | 2,81 | 38 | 4,48 | 0,587 (0,296-1,167) | 0,129 | ||
| rs6534349 | AA | 279 | 71,17 | 665 | 78,33 | 0,006 | Referencia | – |
| AG | 98 | 25,00 | 170 | 20,02 | 1,374 (1,033-1,828) | 0,029 | ||
| GG | 15 | 3,83 | 14 | 1,65 | 2,554 (1,216-5,361) | 0,013 | ||
| rs2069762 | GG | 41 | 10,46 | 78 | 9,19 | 0,545 | Referencia | − |
| GT | 153 | 39,03 | 357 | 42,05 | 0,815 (0,534-1,245) | 0,344 | ||
| TT | 198 | 50,51 | 414 | 48,76 | 0,910 (0,601-1,377) | 0,655 | ||
| rs3136534 | AA | 176 | 44,90 | 346 | 40,75 | 0,223 | Referencia | − |
| AC | 180 | 45,92 | 402 | 47,35 | 0,880 (0,684-1,133) | 0,322 | ||
| CC | 36 | 9,18 | 101 | 11,90 | 0,701 (0,460-1,068) | 0,098 | ||
| rs2243250 | TT | 165 | 42,09 | 333 | 39,22 | 0,536 | Referencia | – |
| TC | 177 | 45,15 | 412 | 48,53 | 0,867 (0,671-1,121) | 0,276 | ||
| CC | 50 | 12,76 | 104 | 12,25 | 0,970 (0,660-1,427) | 0,878 | ||
| rs2070874 | TT | 167 | 42,60 | 337 | 39,69 | 0,531 | Referencia | − |
| TC | 176 | 44,90 | 410 | 48,29 | 0,866 (0,671-1,119) | 0,272 | ||
| CC | 49 | 12,50 | 102 | 12,02 | 0,969 (0,658-1,429) | 0,875 | ||
| rs2227284 | TT | 225 | 57,40 | 408 | 48,06 | 0,001 | Referencia | − |
| TG | 146 | 37,24 | 353 | 41,58 | 0,750 (0,583-0,965) | 0,026 | ||
| GG | 21 | 5,36 | 88 | 10,37 | 0,433 (0,262-0,716) | 0,001 | ||
| rs2243290 | AA | 174 | 44,39 | 331 | 38,99 | 0,190 | Referencia | − |
| AC | 173 | 44,13 | 416 | 49,00 | 0,791 (0,613-1,021) | 0,072 | ||
| CC | 45 | 11,48 | 102 | 12,01 | 0,839 (0,565-1,247) | 0,386 |
Se observó una asociación de riesgo significativa solo para los polimorfismos rs6534349 de IL2 y rs2227284 de IL4 (tabla 2). Para el polimorfismo rs6534349 de IL2, en el genotipo heterocigótico AG y la variante homocigótica GG, el riesgo se multiplicó por 1,374 (IC95%=1,033-1,828, p=0,029) y 2,54 (IC95%=1,216-5,361, p=0,013), respectivamente. Por otro lado, el genotipo heterocigótico TG del polimorfismo rs2227284 de IL4 presentó una odds ratio de 0,750 (IC95%=0,583-0,965, p=0,026), mientras que el genotipo homocigótico GG tuvo una odds ratio de 0,433 (IC95%=0,262-0,716, p=0,001).
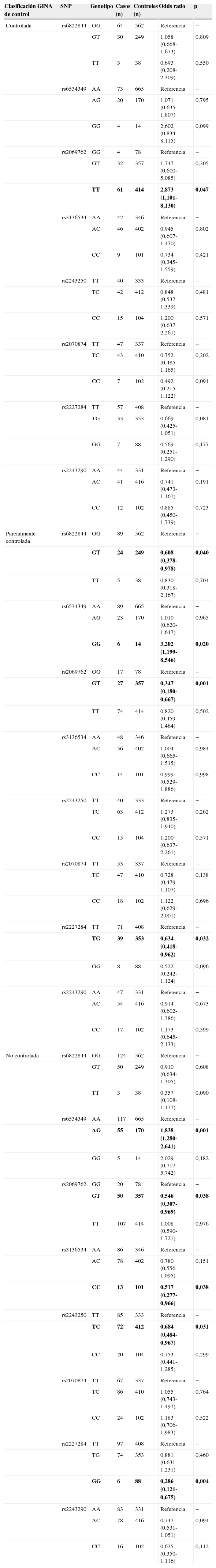
Asociación de riesgo asmático según la clasificación GINA de control del asmaLa distribución genotípica y la asociación de riesgo de los polimorfismos según la clasificación GINA de control del asma se muestran en la tabla 3. En el asma controlada solo halló relación entre el riesgo de padecer la enfermedad y el polimorfismo rs2069762 de IL2 (p=0,047). No se vio que ninguno de los polimorfismos del gen de IL4 aumentara el riesgo ni protegiera a los portadores frente al asma. Por otro lado, en el grupo con asma parcialmente controlada se halló asociación entre el riesgo de sufrir asma y 3 polimorfismos de IL2 (rs6822822, rs2069762, rs6534349) y un polimorfismo de IL4 (rs2227284) (p=0,040, 0,001, 0,020 y 0,032, respectivamente). En el grupo de asma no controlada, 5 polimorfismos mostraron una asociación significativa con el riesgo de padecer asma: rs6534349 (p=0,001), rs2069762 (p=0,038) y rs3136543 (p=0,038) de IL2, así como rs2243250 (p=0,031) y rs2227284 (p=0,004) de IL4.
Distribución genotípica y asociación de riesgo en niños asmáticos y controles sanos, según la clasificación GINA de control del asma
| Clasificación GINA de control | SNP | Genotipo | Casos (n) | Controles (n) | Odds ratio | p |
|---|---|---|---|---|---|---|
| Controlada | rs6822844 | GG | 64 | 562 | Referencia | − |
| GT | 30 | 249 | 1,058 (0,668-1,673) | 0,809 | ||
| TT | 3 | 38 | 0,693 (0,208-2,309) | 0,550 | ||
| rs6534349 | AA | 73 | 665 | Referencia | − | |
| AG | 20 | 170 | 1,071 (0,635-1,807) | 0,795 | ||
| GG | 4 | 14 | 2,602 (0,834-8,115) | 0,099 | ||
| rs2069762 | GG | 4 | 78 | Referencia | − | |
| GT | 32 | 357 | 1,747 (0,600-5,085) | 0,305 | ||
| TT | 61 | 414 | 2,873 (1,101-8,130) | 0,047 | ||
| rs3136534 | AA | 42 | 346 | Referencia | − | |
| AC | 46 | 402 | 0,945 (0,607-1,470) | 0,802 | ||
| CC | 9 | 101 | 0,734 (0,345-1,559) | 0,421 | ||
| rs2243250 | TT | 40 | 333 | Referencia | − | |
| TC | 42 | 412 | 0,848 (0,537-1,339) | 0,481 | ||
| CC | 15 | 104 | 1,200 (0,637-2,261) | 0,571 | ||
| rs2070874 | TT | 47 | 337 | Referencia | − | |
| TC | 43 | 410 | 0,752 (0,485-1,165) | 0,202 | ||
| CC | 7 | 102 | 0,492 (0,215-1,122) | 0,091 | ||
| rs2227284 | TT | 57 | 408 | Referencia | − | |
| TG | 33 | 353 | 0,669 (0,425-1,051) | 0,081 | ||
| GG | 7 | 88 | 0,569 (0,251-1,290) | 0,177 | ||
| rs2243290 | AA | 44 | 331 | Referencia | − | |
| AC | 41 | 416 | 0,741 (0,473-1,161) | 0,191 | ||
| CC | 12 | 102 | 0,885 (0,450-1,739) | 0,723 | ||
| Parcialmente controlada | rs6822844 | GG | 89 | 562 | Referencia | − |
| GT | 24 | 249 | 0,608 (0,378-0,978) | 0,040 | ||
| TT | 5 | 38 | 0,830 (0,318-2,167) | 0,704 | ||
| rs6534349 | AA | 89 | 665 | Referencia | − | |
| AG | 23 | 170 | 1,010 (0,620-1,647) | 0,965 | ||
| GG | 6 | 14 | 3,202 (1,199-8,546) | 0,020 | ||
| rs2069762 | GG | 17 | 78 | Referencia | − | |
| GT | 27 | 357 | 0,347 (0,180-0,667) | 0,001 | ||
| TT | 74 | 414 | 0,820 (0,459-1,464) | 0,502 | ||
| rs3136534 | AA | 48 | 346 | Referencia | − | |
| AC | 56 | 402 | 1,004 (0,665-1,515) | 0,984 | ||
| CC | 14 | 101 | 0,999 (0,529-1,886) | 0,998 | ||
| rs2243250 | TT | 40 | 333 | Referencia | − | |
| TC | 63 | 412 | 1,273 (0,835-1,940) | 0,262 | ||
| CC | 15 | 104 | 1,200 (0,637-2,261) | 0,571 | ||
| rs2070874 | TT | 53 | 337 | Referencia | − | |
| TC | 47 | 410 | 0,728 (0,479-1,107) | 0,138 | ||
| CC | 18 | 102 | 1,122 (0,629-2,001) | 0,696 | ||
| rs2227284 | TT | 71 | 408 | Referencia | − | |
| TG | 39 | 353 | 0,634 (0,418-0,962) | 0,032 | ||
| GG | 8 | 88 | 0,522 (0,242-1,124) | 0,096 | ||
| rs2243290 | AA | 47 | 331 | Referencia | − | |
| AC | 54 | 416 | 0,914 (0,602-1,386) | 0,673 | ||
| CC | 17 | 102 | 1,173 (0,645-2,133) | 0,599 | ||
| No controlada | rs6822844 | GG | 124 | 562 | Referencia | − |
| GT | 50 | 249 | 0,910 (0,634-1,305) | 0,608 | ||
| TT | 3 | 38 | 0,357 (0,108-1,177) | 0,090 | ||
| rs6534349 | AA | 117 | 665 | Referencia | − | |
| AG | 55 | 170 | 1,838 (1,280-2,641) | 0,001 | ||
| GG | 5 | 14 | 2,029 (0,717-5,742) | 0,182 | ||
| rs2069762 | GG | 20 | 78 | Referencia | − | |
| GT | 50 | 357 | 0,546 (0,307-0,969) | 0,038 | ||
| TT | 107 | 414 | 1,008 (0,590-1,721) | 0,976 | ||
| rs3136534 | AA | 86 | 346 | Referencia | − | |
| AC | 78 | 402 | 0,780 (0,556-1,095) | 0,151 | ||
| CC | 13 | 101 | 0,517 (0,277-0,966) | 0,038 | ||
| rs2243250 | TT | 85 | 333 | Referencia | − | |
| TC | 72 | 412 | 0,684 (0,484-0,967) | 0,031 | ||
| CC | 20 | 104 | 0,753 (0,441-1,285) | 0,299 | ||
| rs2070874 | TT | 67 | 337 | Referencia | − | |
| TC | 86 | 410 | 1,055 (0,743-1,497) | 0,764 | ||
| CC | 24 | 102 | 1,183 (0,706-1,983) | 0,522 | ||
| rs2227284 | TT | 97 | 408 | Referencia | − | |
| TG | 74 | 353 | 0,881 (0,631-1,231) | 0,460 | ||
| GG | 6 | 88 | 0,286 (0,121-0,675) | 0,004 | ||
| rs2243290 | AA | 83 | 331 | Referencia | − | |
| AC | 78 | 416 | 0,747 (0,531-1,051) | 0,094 | ||
| CC | 16 | 102 | 0,625 (0,350-1,116) | 0,112 |
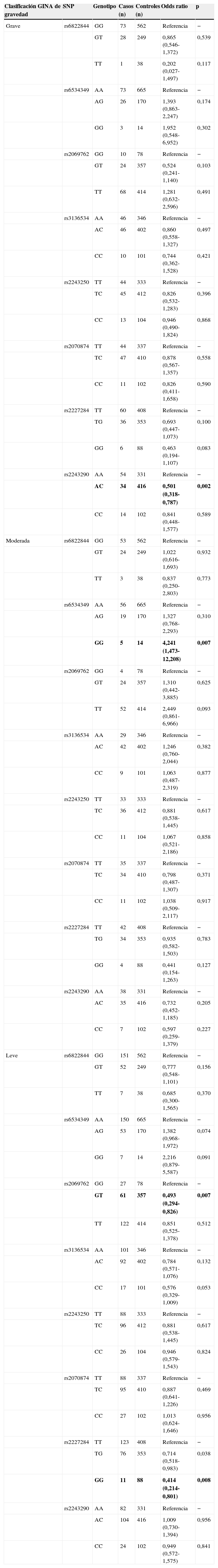
La distribución de los genotipos de los polimorfismos en pacientes con distintos niveles de gravedad del asma se muestra en la tabla 4, junto con la asociación de riesgo correspondiente. En los sujetos con asma grave se observaron asociaciones significativas con el rs2243290 (p=0,002). En el caso del asma moderada, solo el rs6534349 fue significativo (p=0,007). Además, en el asma leve la asociación de riesgo estaba presente en los polimorfismos rs2069762 y rs2227284 (p=0,007 y p=0,008, respectivamente).
Distribución genotípica y asociación de riesgo en niños asmáticos y controles sanos, según la clasificación GINA de gravedad del asma
| Clasificación GINA de gravedad | SNP | Genotipo | Casos (n) | Controles (n) | Odds ratio | p |
|---|---|---|---|---|---|---|
| Grave | rs6822844 | GG | 73 | 562 | Referencia | − |
| GT | 28 | 249 | 0,865 (0,546-1,372) | 0,539 | ||
| TT | 1 | 38 | 0,202 (0,027-1,497) | 0,117 | ||
| rs6534349 | AA | 73 | 665 | Referencia | − | |
| AG | 26 | 170 | 1,393 (0,863-2,247) | 0,174 | ||
| GG | 3 | 14 | 1,952 (0,548-6,952) | 0,302 | ||
| rs2069762 | GG | 10 | 78 | Referencia | − | |
| GT | 24 | 357 | 0,524 (0,241-1,140) | 0,103 | ||
| TT | 68 | 414 | 1,281 (0,632-2,596) | 0,491 | ||
| rs3136534 | AA | 46 | 346 | Referencia | − | |
| AC | 46 | 402 | 0,860 (0,558-1,327) | 0,497 | ||
| CC | 10 | 101 | 0,744 (0,362-1,528) | 0,421 | ||
| rs2243250 | TT | 44 | 333 | Referencia | − | |
| TC | 45 | 412 | 0,826 (0,532-1,283) | 0,396 | ||
| CC | 13 | 104 | 0,946 (0,490-1,824) | 0,868 | ||
| rs2070874 | TT | 44 | 337 | Referencia | − | |
| TC | 47 | 410 | 0,878 (0,567-1,357) | 0,558 | ||
| CC | 11 | 102 | 0,826 (0,411-1,658) | 0,590 | ||
| rs2227284 | TT | 60 | 408 | Referencia | − | |
| TG | 36 | 353 | 0,693 (0,447-1,073) | 0,100 | ||
| GG | 6 | 88 | 0,463 (0,194-1,107) | 0,083 | ||
| rs2243290 | AA | 54 | 331 | Referencia | − | |
| AC | 34 | 416 | 0,501 (0,318-0,787) | 0,002 | ||
| CC | 14 | 102 | 0,841 (0,448-1,577) | 0,589 | ||
| Moderada | rs6822844 | GG | 53 | 562 | Referencia | − |
| GT | 24 | 249 | 1,022 (0,616-1,693) | 0,932 | ||
| TT | 3 | 38 | 0,837 (0,250-2,803) | 0,773 | ||
| rs6534349 | AA | 56 | 665 | Referencia | − | |
| AG | 19 | 170 | 1,327 (0,768-2,293) | 0,310 | ||
| GG | 5 | 14 | 4,241 (1,473-12,208) | 0,007 | ||
| rs2069762 | GG | 4 | 78 | Referencia | − | |
| GT | 24 | 357 | 1,310 (0,442-3,885) | 0,625 | ||
| TT | 52 | 414 | 2,449 (0,861-6,966) | 0,093 | ||
| rs3136534 | AA | 29 | 346 | Referencia | − | |
| AC | 42 | 402 | 1,246 (0,760-2,044) | 0,382 | ||
| CC | 9 | 101 | 1,063 (0,487-2,319) | 0,877 | ||
| rs2243250 | TT | 33 | 333 | Referencia | − | |
| TC | 36 | 412 | 0,881 (0,538-1,445) | 0,617 | ||
| CC | 11 | 104 | 1,067 (0,521-2,186) | 0,858 | ||
| rs2070874 | TT | 35 | 337 | Referencia | − | |
| TC | 34 | 410 | 0,798 (0,487-1,307) | 0,371 | ||
| CC | 11 | 102 | 1,038 (0,509-2,117) | 0,917 | ||
| rs2227284 | TT | 42 | 408 | Referencia | − | |
| TG | 34 | 353 | 0,935 (0,582-1,503) | 0,783 | ||
| GG | 4 | 88 | 0,441 (0,154-1,263) | 0,127 | ||
| rs2243290 | AA | 38 | 331 | Referencia | − | |
| AC | 35 | 416 | 0,732 (0,452-1,185) | 0,205 | ||
| CC | 7 | 102 | 0,597 (0,259-1,379) | 0,227 | ||
| Leve | rs6822844 | GG | 151 | 562 | Referencia | − |
| GT | 52 | 249 | 0,777 (0,548-1,101) | 0,156 | ||
| TT | 7 | 38 | 0,685 (0,300-1,565) | 0,370 | ||
| rs6534349 | AA | 150 | 665 | Referencia | − | |
| AG | 53 | 170 | 1,382 (0,968-1,972) | 0,074 | ||
| GG | 7 | 14 | 2,216 (0,879-5,587) | 0,091 | ||
| rs2069762 | GG | 27 | 78 | Referencia | − | |
| GT | 61 | 357 | 0,493 (0,294-0,826) | 0,007 | ||
| TT | 122 | 414 | 0,851 (0,525-1,378) | 0,512 | ||
| rs3136534 | AA | 101 | 346 | Referencia | − | |
| AC | 92 | 402 | 0,784 (0,571-1,076) | 0,132 | ||
| CC | 17 | 101 | 0,576 (0,329-1,009) | 0,053 | ||
| rs2243250 | TT | 88 | 333 | Referencia | − | |
| TC | 96 | 412 | 0,881 (0,538-1,445) | 0,617 | ||
| CC | 26 | 104 | 0,946 (0,579-1,543) | 0,824 | ||
| rs2070874 | TT | 88 | 337 | Referencia | − | |
| TC | 95 | 410 | 0,887 (0,641-1,226) | 0,469 | ||
| CC | 27 | 102 | 1,013 (0,624-1,646) | 0,956 | ||
| rs2227284 | TT | 123 | 408 | Referencia | − | |
| TG | 76 | 353 | 0,714 (0,518-0,983) | 0,038 | ||
| GG | 11 | 88 | 0,414 (0,214-0,801) | 0,008 | ||
| rs2243290 | AA | 82 | 331 | Referencia | − | |
| AC | 104 | 416 | 1,009 (0,730-1,394) | 0,956 | ||
| CC | 24 | 102 | 0,949 (0,572-1,575) | 0,841 |
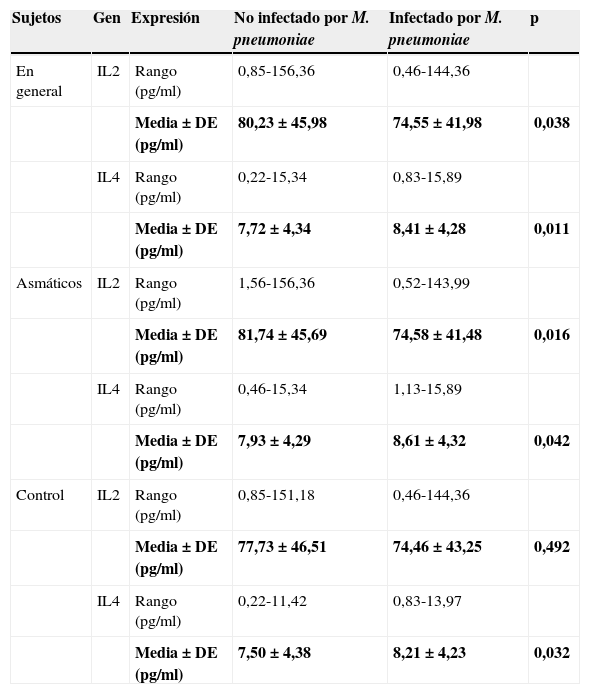
La tabla 5 muestra los niveles séricos de expresión (en cuanto a concentración) de IL-2 e IL-4 en sujetos no infectados e infectados por M.pneumoniae. La concentración promedio de IL-2 en sujetos no infectados por M.pneumoniae fue significativamente mayor que en los sujetos infectados por M.pneumoniae (p=0,038). En el caso de la IL-4 se observó lo contrario (p=0,011).
Nivel sérico de expresión de IL-2 e IL-4 en sujetos infectados y no infectados por Mycoplasma pneumoniae
| Sujetos | Gen | Expresión | No infectado por M. pneumoniae | Infectado por M. pneumoniae | p |
|---|---|---|---|---|---|
| En general | IL2 | Rango (pg/ml) | 0,85-156,36 | 0,46-144,36 | |
| Media±DE (pg/ml) | 80,23±45,98 | 74,55±41,98 | 0,038 | ||
| IL4 | Rango (pg/ml) | 0,22-15,34 | 0,83-15,89 | ||
| Media±DE (pg/ml) | 7,72±4,34 | 8,41±4,28 | 0,011 | ||
| Asmáticos | IL2 | Rango (pg/ml) | 1,56-156,36 | 0,52-143,99 | |
| Media±DE (pg/ml) | 81,74±45,69 | 74,58±41,48 | 0,016 | ||
| IL4 | Rango (pg/ml) | 0,46-15,34 | 1,13-15,89 | ||
| Media±DE (pg/ml) | 7,93±4,29 | 8,61±4,32 | 0,042 | ||
| Control | IL2 | Rango (pg/ml) | 0,85-151,18 | 0,46-144,36 | |
| Media±DE (pg/ml) | 77,73±46,51 | 74,46±43,25 | 0,492 | ||
| IL4 | Rango (pg/ml) | 0,22-11,42 | 0,83-13,97 | ||
| Media±DE (pg/ml) | 7,50±4,38 | 8,21±4,23 | 0,032 |
Cuando se clasificaron los sujetos en función de la presencia de asma, se halló una tendencia similar en los pacientes asmáticos (p=0,016 para IL-2; p=0,042 para IL-4). Sin embargo, en los controles no asmáticos, los niveles de IL-2 entre los sujetos infectados y no infectados por M.pneumoniae no difirieron significativamente (p=0,492). No obstante, al igual que los sujetos asmáticos, se detectó un promedio de concentración de IL-4 significativamente menor entre los pacientes no infectados por M.pneumoniae en comparación con los infectados por M. pneumoniae (p=0,032).
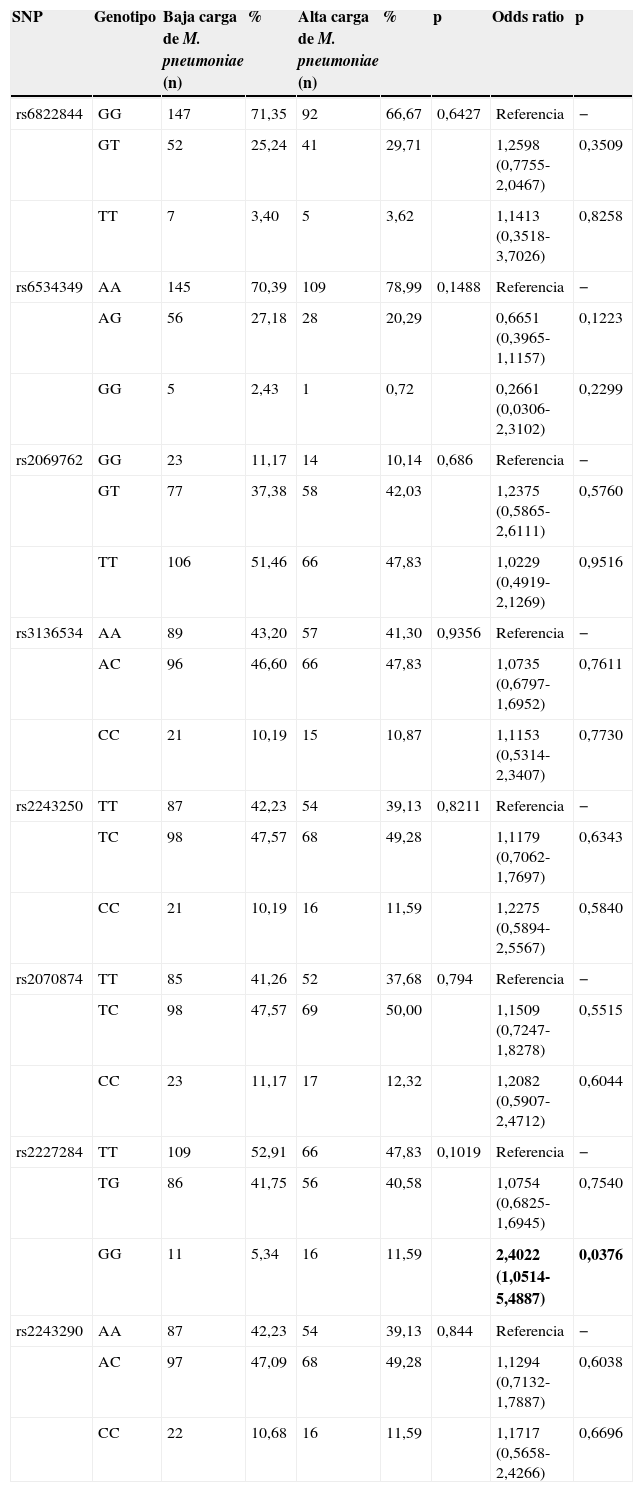
Distribución genotípica en pacientes infectados por Mycoplasma pneumoniae con infecciones de carga baja y altaLa tabla 6 resume la distribución genotípica de los 8 polimorfismos en los sujetos infectados por M.pneumoniae con cargas bacterianas bajas y altas. No se observó una diferencia significativa entre los 2 grupos de sujetos del estudio en cuanto a distribución genotípica de los 8 polimorfismos (p>0,05). Ninguna de las distribuciones genotípicas se desvió significativamente del equilibrio de Hardy-Weinberg (p>0,05).
Distribución genotípica y asociación de riesgo en todos los sujetos con cargas bajas y altas de Mycoplasma pneumoniae
| SNP | Genotipo | Baja carga de M. pneumoniae (n) | % | Alta carga de M. pneumoniae (n) | % | p | Odds ratio | p |
|---|---|---|---|---|---|---|---|---|
| rs6822844 | GG | 147 | 71,35 | 92 | 66,67 | 0,6427 | Referencia | − |
| GT | 52 | 25,24 | 41 | 29,71 | 1,2598 (0,7755-2,0467) | 0,3509 | ||
| TT | 7 | 3,40 | 5 | 3,62 | 1,1413 (0,3518-3,7026) | 0,8258 | ||
| rs6534349 | AA | 145 | 70,39 | 109 | 78,99 | 0,1488 | Referencia | − |
| AG | 56 | 27,18 | 28 | 20,29 | 0,6651 (0,3965-1,1157) | 0,1223 | ||
| GG | 5 | 2,43 | 1 | 0,72 | 0,2661 (0,0306-2,3102) | 0,2299 | ||
| rs2069762 | GG | 23 | 11,17 | 14 | 10,14 | 0,686 | Referencia | − |
| GT | 77 | 37,38 | 58 | 42,03 | 1,2375 (0,5865-2,6111) | 0,5760 | ||
| TT | 106 | 51,46 | 66 | 47,83 | 1,0229 (0,4919-2,1269) | 0,9516 | ||
| rs3136534 | AA | 89 | 43,20 | 57 | 41,30 | 0,9356 | Referencia | − |
| AC | 96 | 46,60 | 66 | 47,83 | 1,0735 (0,6797-1,6952) | 0,7611 | ||
| CC | 21 | 10,19 | 15 | 10,87 | 1,1153 (0,5314-2,3407) | 0,7730 | ||
| rs2243250 | TT | 87 | 42,23 | 54 | 39,13 | 0,8211 | Referencia | − |
| TC | 98 | 47,57 | 68 | 49,28 | 1,1179 (0,7062-1,7697) | 0,6343 | ||
| CC | 21 | 10,19 | 16 | 11,59 | 1,2275 (0,5894-2,5567) | 0,5840 | ||
| rs2070874 | TT | 85 | 41,26 | 52 | 37,68 | 0,794 | Referencia | − |
| TC | 98 | 47,57 | 69 | 50,00 | 1,1509 (0,7247-1,8278) | 0,5515 | ||
| CC | 23 | 11,17 | 17 | 12,32 | 1,2082 (0,5907-2,4712) | 0,6044 | ||
| rs2227284 | TT | 109 | 52,91 | 66 | 47,83 | 0,1019 | Referencia | − |
| TG | 86 | 41,75 | 56 | 40,58 | 1,0754 (0,6825-1,6945) | 0,7540 | ||
| GG | 11 | 5,34 | 16 | 11,59 | 2,4022 (1,0514-5,4887) | 0,0376 | ||
| rs2243290 | AA | 87 | 42,23 | 54 | 39,13 | 0,844 | Referencia | − |
| AC | 97 | 47,09 | 68 | 49,28 | 1,1294 (0,7132-1,7887) | 0,6038 | ||
| CC | 22 | 10,68 | 16 | 11,59 | 1,1717 (0,5658-2,4266) | 0,6696 |
La relación entre los 8 polimorfismos y el riesgo de predisposición a infecciones con alta carga de M.pneumoniae también se muestra en la tabla 6. Solo se observó una relación significativa en el polimorfismo rs2227284 de IL4 (p=0,0376). No se halló ninguna relación estadísticamente significativa en el resto de polimorfismos (p>0,05).
DiscusiónEl asma es una enfermedad de las vías respiratorias que se debe principalmente a la inflamación crónica del sistema respiratorio. Se considera que la infección por M.pneumoniae fomenta la exacerbación de los síntomas asmáticos5,6. Las citoquinas son mediadores clave en las reacciones inmunitarias e inflamatorias, por lo que podrían desempeñar un papel importante en la aparición del asma y la erradicación de la infección por M.pneumoniae. La IL-2 y la IL-4 son 2 citoquinas importantes que se han vinculado al desarrollo de asma. Partimos de la hipótesis de que los polimorfismos en los genes de IL2 e IL4 podrían estar relacionados con el riesgo de padecer asma e infección por M.pneumoniae en niños.
Hemos investigado la relación entre 4 polimorfismos de IL2 y 4 polimorfismos de IL4 y el riesgo de padecer asma en niños. Nuestros resultados demostraron que el polimorfismo rs6534349 de IL2 y el polimorfismo rs2227284 de IL4 podían aumentar y reducir, respectivamente, el riesgo de padecer asma de forma significativa. Esta relación dependía de la dosis. En otras palabras, el riesgo y los efectos protectores de los polimorfismos fueron mayores cuando los alelos variantes estaban presentes en 2 copias (variante homocigótica) que cuando estaban solo en una copia (heterocigótico). Además, también hemos analizado la relación entre los polimorfismos con distintos niveles de control y gravedad del asma, conforme a la clasificación GINA. Hemos descubierto que entre los pacientes del grupo «controlada», solo un polimorfismo (rs2069762) se asociaba al riesgo de padecer asma, mientras que en el grupo «parcialmente controlada» fueron 4 polimorfismos (rs6822844, rs6534349, rs2069762 y rs2227284) y en el grupo «no controlada» fueron 5 polimorfismos (rs6534349, rs2069762, rs3136534, rs2243250 y rs2227284). El aumento del número de polimorfismos asociados al riesgo de padecer asma y el incremento de la pérdida de control sugieren la implicación de una amplia red de interacciones genéticas, lo que resalta la complejidad de la afección.
De todos modos, el número de polimorfismos significativamente relacionados con el riesgo de padecer asma fue similar en pacientes de distintos niveles de gravedad. Los pacientes con asma leve presentaron asociación de riesgo con 2 polimorfismos (rs2069762 y rs2227284), mientras que los pacientes con asma moderada o grave tuvieron asociación con un polimorfismo cada uno (rs6534349 en moderada, rs2243290 en grave). Es interesante resaltar que los pacientes asmáticos con distintos grados de gravedad presentaban polimorfismos totalmente diferentes. Esto sugiere que cada polimorfismo podría ejercer un efecto único y exclusivo que contribuye a la aparición del asma. Sin embargo, cabe mencionar también que cuando se realizó el análisis conforme a los niveles de gravedad del asma, el tamaño muestral fue demasiado bajo como para garantizar una interpretación fiable de los datos14. Esta cuestión es una de las limitaciones del presente estudio.
Nuestra propuesta es que estos polimorfismos podrían alterar la expresión de los productos proteicos, lo que causaría las modificaciones del riesgo explicadas más arriba15. En concreto, postulamos que los alelos variantes de los polimorfismos rs6822844, rs2069762 y rs3136534 podrían reducir la expresión de los productos proteicos, mientras que los de los polimorfismos rs6534349, rs2243250, rs2227284 y rs2243290 podrían aumentar la expresión de los mismos. Nuestro postulado se basa en el hecho de que la IL-2 es una citoquina proinflamatoria, por lo que una mayor expresión de IL-2 puede conllevar un mayor nivel de inflamación, lo que facilita el desarrollo de asma, y viceversa16. Por el contrario, la IL-4 es una proteína antiinflamatoria, de modo que un aumento de la expresión de IL-4 podría conllevar una reducción del nivel de inflamación, lo que, a su vez, protegería al anfitrión frente al asma, y viceversa. No obstante, se precisan más estudios experimentales para confirmar nuestros postulados. También observamos que no existía relación entre el polimorfismo rs2070874 y el riesgo de padecer asma en todas las clasificaciones de los sujetos del estudio (es decir, clasificación basada en control según GINA y clasificación basada en la gravedad según GINA). Esto sugiere que el polimorfismo no participa en la aparición del asma.
También estudiamos la diferencia en los niveles séricos de expresión de IL-2 e IL-4 entre sujetos con y sin infección por M.pneumoniae. Encontramos una expresión significativamente mayor de IL-2 e IL-4 en los pacientes no infectados por M.pneumoniae e infectados por M.pneumoniae, respectivamente. No se trata de un hallazgo inesperado, ya que la inflamación es una de las primeras respuestas del sistema inmunitario17. Suponemos que un nivel de IL-2 mayor, que estimula la inflamación, es importante a la hora de rechazar las infecciones patógenas18. Los sujetos con mayor nivel de IL-2 fueron, por lo tanto, los que no estaban infectados por M.pneumoniae. La relación entre los niveles de IL-2 y la infección por M.pneumoniae fue significativa solo entre los pacientes asmáticos, pero no en los controles. Esta observación coincide con el hecho de que la IL-2 desempeña un papel importante en la patogenia del asma19. Por su parte, la IL-4 suprime la inflamación, de modo que un alto nivel de IL-4 no permite al sistema inmunitario del anfitrión funcionar de forma óptima. Los pacientes que presentaban un nivel mayor de IL-4 fueron, por lo tanto, los que estaban infectados por M.pneumoniae, independientemente de si se trataba de pacientes asmáticos o controles no asmáticos.
Las enfermedades respiratorias más graves en términos clínicos presentaban cargas mayores de infección por M.pneumoniae20. Por eso, seguimos comparando la distribución de los 8 polimorfismos de IL2 e IL4 en sujetos con alta y baja carga de M.pneumoniae con el objetivo de identificar si alguno de los polimorfismos está relacionado con infección con baja carga del patógeno. No se hallaron diferencias significativas en la distribución de los polimorfismos entre ambos grupos, pero se observó una asociación de riesgo significativa en el genotipo de variante homocigótica del polimorfismo rs2227284. Esto sugiere que el genotipo de variante homocigótica del polimorfismo rs2227284 podría predisponer a sus portadores a infecciones con alta carga de M.pneumoniae, tal y como explicaban Wang et al.21 cuando demostraron que el polimorfismo rs2227284 podía afectar a la respuesta humoral.
De hecho, varios estudios han investigado la relación entre distintos polimorfismos en los genes de IL2 e IL4 y el riesgo de padecer asma22-24. No obstante, la mayoría de dichos estudios solo incluían uno o 2 polimorfismos en su análisis, lo que podría ocasionar interpretaciones engañosas, ya que el efecto de un polimorfismo podría compensarse con la presencia de otros polimorfismos en el mismo gen25. La fuerza de nuestro estudio radica en la incorporación de un gran número de polimorfismos en 2 genes estrechamente relacionados.
ConclusionesEn este estudio identificamos 2 polimorfismos que podrían usarse como biomarcadores predictivos para calcular el riesgo de padecer asma en niños. También hemos demostrado que los niveles séricos de IL-2 e IL-4 diferían significativamente entre los sujetos con y sin infección por M.pneumoniae. Entre los sujetos infectados por M.pneumoniae demostramos que el genotipo GG rs2227284 presentaba mayores probabilidades de sufrir una infección con alta carga. Sin embargo, reconocemos que existen varias limitaciones en el presente estudio, como el pequeño tamaño muestral, especialmente por lo que respecta a sujetos con infección por M.pneumoniae. En este sentido, se necesitan estudios adicionales con un mayor tamaño muestral.
FinanciaciónEste estudio ha sido financiado por los autores.
AutoríaRSW y HXJ seleccionaron a los sujetos del estudio, extrajeron el ADN de todas las muestras y realizaron la detección de la infección por Mycoplasma pneumoniae. SQS genotipó los SNP y redactó el artículo. XYL y SJC cuantificaron los niveles séricos de expresión de IL-2 e IL-4 y realizaron los análisis estadísticos. ZBJ ideó el estudio y participó en su diseño y coordinación, y también ayudó a redactar el artículo. Todos los autores han leído y dado su aprobación al artículo.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses, ni directo ni indirecto, respecto al contenido del artículo.
Agradecemos a los enfermeros de nuestros hospitales por su ayuda a la hora de seleccionar participantes.