La hipertensión arterial pulmonar es una enfermedad infrecuente caracterizada por una obstrucción progresiva de las arterias pulmonares. El receptor tipo II de la proteína ósea morfogenética (BMPR2) es el principal gen relacionado con esta enfermedad. EL objetivo de este trabajo fue analizar el patrón de metilación de la región promotora del gen BMPR2 en individuos con hipertensión arterial pulmonar y controles.
MétodosSe analizó la región a estudio con los softwares Methyl Primer Express® v.1.0 y MatInspector. El ADN genómico, obtenido a partir de sangre periférica, fue modificado con bisulfito de sodio. La metilación se analizó utilizando PCR específica de metilación. Como control positivo de metilación se utilizó ADN tratado con CpG metiltransferasa y como control positivo de expresión se utilizó ADN del cultivo celular H1299.
ResultadosSe ha identificado una isla CpG susceptible de presentar metilación en la región promotora del gen que contiene secuencias específicas como sitios de unión a factores de trascripción NIT-2 (global-acting regulatory protein), sex-determining region Y y heat shock factor. No se han encontrado indicios de metilación en los pacientes ni en los controles. En las células H1299, que expresan el gen BMPR2, no se ha identificado metilación en la isla CpG analizada.
ConclusionesNo se detectaron indicios de metilación en la región promotora a estudio en pacientes y controles, siendo esta la región más adecuada para realizar el estudio debido al alto número de sitios de unión a factores de transcripción que podrían estar involucrados en la correcta funcionalidad del gen.
Pulmonary arterial hypertension is characterizated by obstruction of the pulmonary arteries. The gene mainly related to pathology is the bone morphogenetic protein receptor type II (BMPR2). The aim of this study was to analyze the methylation pattern of the BMPR2 promoter region in patients and controls.
MethodsWe used Methyl Primer Express® v.1.0 and MatInspector softwares to analyze this region. Genomic DNA obtained from the peripheral blood of patients and controls was modified with sodium bisulphite. Methylation was analyzed using methylation-specific PCR. DNA treated with CpG methyltransferase was used as a positive control for methylation and H1299 cell culture DNA was used as positive control for gene expression.
ResultsWe identified a CpG island, which may have been methylated, in the BMPR2 promoter region, in addition to NIT-2 (global-acting regulatory protein), sex-determining region Y) and heat shock factor transcription factor binding sites. We found no evidence of methylation in patients and controls. No methylated CpG sites were identified in H1299 cells expressing the BMPR2 gene.
ConclusionsThe BMPR2 promoter region is the most suitable for study because of the high number of transcription factor binding sites that could alter gene function. No evidence of methylation was detected in this region in patients and controls.
La hipertensión arterial pulmonar (HAP; OMIM #178600; ORPHA 422) es una enfermedad progresiva que se caracteriza por la obstrucción de las arterias pulmonares1. Se define por un aumento sostenido de la presión arterial pulmonar media (PAPm) ≥ 25mmHg en reposo2. Los síntomas incluyen disnea, síncope, dolor torácico y, finalmente, la muerte prematura por insuficiencia cardiaca derecha1. Los cambios estructurales y funcionales en la pared vascular y la formación de trombos son los principales factores responsables del aumento de la resistencia vascular pulmonar (RVP)3. La HAP puede ser hereditaria (HAPF), idiopática (HAPI) o asociada a otras enfermedades, medicamentos o tóxicos (HAPA)4. La enfermedad es más frecuente en mujeres, con un ratio de 1,7:1 respecto a varones2.
Hasta el momento, la base genética de la HAP se ha relacionado fundamentalmente con mutaciones en el gen que codifica para el receptor tipo II de la proteína ósea morfogenética (BMPR2). Este gen se localiza en el cromosoma 2q33 y se han identificado mutaciones en más del 80% de los pacientes con HAPF. Sin embargo, solo el 20% de los portadores de mutaciones en este gen desarrollan la enfermedad5,6. En pacientes con HAPI, la frecuencia de mutaciones en el gen BMPR2 es menor, encontrándose en pequeñas cohortes de pacientes entre el 6-40%6–9. Desde la identificación de este gen como principal causa de desarrollo de HAP se han descrito diferentes genes implicados en la enfermedad9 y algunos moduladores genéticos como el gen SLC6A410, EDN111, NOS212, AGTR1 y TRPC613 capaces de modificar el fenotipo de los pacientes.
La metilación del ADN es una modificación que está presente en numerosos organismos14–17. La metilación de la región promotora de un gen provoca el silenciamiento transcripcional del mismo15,18,19. Los promotores asociados a estas islas CpG muestran múltiples sitios de inicio de la transcripción dispersos a lo largo de la isla20 y funcionan frecuentemente como promotores bidireccionales21–23.
Las proteínas BMP son moléculas de señalización que pertenecen a la superfamilia del TGF-β (factor de crecimiento transformante beta)24,25. Tienen un importante papel en la regulación de la diferenciación celular, la proliferación y la apoptosis. La pérdida de función o la reducción de la expresión de BMPR2 puede ser suficiente para el desarrollo de HAP24,25.
La región promotora del gen BMPR2 presenta una secuencia codificante conservada que posee un especial interés en la regulación génica. Estas secuencias codificantes conservadas están relacionadas con los sitios de unión de factores de transcripción y otros elementos reguladores que actúan en cis. Por ese motivo, el objetivo de este trabajo fue analizar el patrón de metilación de la isla CpG localizada en los últimos 2.000 pb de la región promotora del gen BMPR2, en concreto en la zona de esta isla donde se encuentra la secuencia de unión de un mayor número de factores de transcripción, en ADN de sangre periférica de pacientes con HAPI y HAPA para investigar si los cambios encontrados producen algún cambio en la penetrancia de la enfermedad.
Pacientes y métodosProcedencia y evaluación clínica de los pacientesLos pacientes objeto de estudio se reclutaron en hospitales de Galicia, Hospital Valdecilla de Santander y de la Asociación Española de Hipertensión Pulmonar. En todos los casos se obtuvo el consentimiento informado, actuándose de acuerdo con los principios éticos para la investigación médica en seres humanos establecidos en la declaración de Helsinki (http://www.wma.net) y promulgados por la Asociación Médica Mundial. El Comité Ético local (Comité Autonómico de Ética da Investigación de Galicia) dio su aprobación para el estudio.
La selección de los pacientes fue consecutiva, tanto en los hospitales como en la Asociación Española de Hipertensión Pulmonar, en base a cumplir los criterios de inclusión y aceptar participar en el estudio. Los pacientes fueron diagnosticados y clasificados en base a su historia clínica, así como en función del resultado de diversas pruebas hemodinámicas. El diagnóstico de HAP se basó en un cateterismo cardiaco derecho con una PAPm ≥25mmHg y una presión arterial de enclavamiento ≤ a 15mmHg sin tratamiento específico. En todos los casos se siguió el protocolo de manejo habitual acorde a las recomendaciones de la European Respiratory Society/European Society of Cardiology26. Se recogieron datos clínicos como clase funcional (CF), test de 6 minutos de marcha, función pulmonar y estudios de laboratorio. Las variables hemodinámicas incluidas fueron la PAPm, presión arterial pulmonar sistólica, presión arterial de enclavamiento, índice cardiaco, resistencias vasculares pulmonares y prueba de vasorreactividad. Los pacientes se encontraban estables en el momento de la realización del cateterismo.
Para este estudio se han utilizado muestras de 50 individuos sanos sin patología respiratoria aparente elegidos de la población general para la determinación de la existencia de variabilidad en cuanto a metilación, estas muestras fueron recogidas en el Complexo Hospitalario Universitario de Vigo.
Búsqueda y análisis de islas CpG en el gen BMPR2Con el programa Methyl Primer Express® Version 1.0 se detectaron las islas CpG de la región promotora del gen BMPR2 utilizando la secuencia de referencia ENST00000374580 de ENSEMBL. Después de insertar la secuencia de ADN seleccionada en el programa, este detecta todas las islas CpG posibles y ofrece diseños de cebadores específicos basados en las secuencias diana seleccionadas. Metil Primer Software Express® permite diseñar cebadores para la amplificación mediante PCR específica de metilación (MSP), una de las técnicas más utilizadas para la cartografía de la metilación.
Mediante el programa MatInspectorn, de Genomatix, se analizaron las regiones elegidas para comprobar la existencia de elementos reguladores de la transcripción y los sitios de unión.
Análisis de la metilaciónLas muestras de ADN se trataron con bisulfito de sodio usando el kit EZ DNA Methylation-Direct™ KIT (Zymo Research, Irvine, CA, EE. UU.) según las indicaciones del fabricante. Para resultados óptimos se recomienda una cantidad de ADN de partida entre 200-500ng. En cada tratamiento de bisulfito de las muestras de ADN provenientes de sangre periférica de pacientes y controles se añadió un control negativo (ADN sin tratar) y un control positivo tratado con CpG metiltransferasa (MSssI), de New England Biolabs (Ipswich, MA, EE. UU.). Tanto el control positivo como el control negativo procedían de un mismo individuo sano. Para el control positivo se mezcló 34.5μL de H2O estéril, 4μL de tampón sin Mg2+, 4μL de SAM 1/10, 1,8μL de M.SsssI y 6μL de ADN incubándose en un baño de agua a 37°C durante 16 horas. Tras la modificación mediante bisulfito se cuantificó el ADN total de las muestras, mediante el espectrofotómetro NanoDrop 2000c Spectrophotometer (Thermo Scientific, Waltham, MA, EE. UU.).
Para la PCR específica de metilación (MSP) se utilizó 1-4μL de ADN modificado con bisulfito. La amplificación de los fragmentos se realizó mediante la taq TaKaRa Ex Taq Hot Start Version (TAKARA BIO INC, Otsu, Shiga, Japón) siguiendo las indicaciones del fabricante. El volumen final de la reacción de PCR se ajustó con H2O estéril hasta 25μL. Se prepararon 2 Mix de PCR, uno con los cebadores metilados y otro con los cebadores no metilados (tabla 1). Las condiciones de la PCR fueron: 5 minutos a 95°C, 38 ciclos de 30 segundos a 95°C, 30 segundos a 60°C y 30 segundos a 72°C, terminando con una extensión final a 72°C durante 7 minutos.
Cebadores empleados para la amplificación de las MSP de la región promotora del gen BMPR2
| Amplicón | Metilados | No metilados | Temperatura y tamaño |
|---|---|---|---|
| 1 | F: CGTAGTGCGGGTTTTATTTC R: GCAACTACCAATCTACGACG | F: AGGTGTAGTGTGGGTTTTATTTT R: ACAACTACCAATCTACAACACTC | 60°C y 170pb |
| 2 | F: GAGGGACGTAGGGTGTTTC R: TCCTAACGAAACTCGAACGT | F: GGAGGGATGTAGGGTGTTTT R: CTCCTAACAAAACTCAAACAT | 60°C y 112pb |
| 3 | F: GTTTAGAGTTGCGGGAGAAC R: TTCCTACGACTCGATCTTACC | F: GGAGTTTAGAGTTGTGGGAGAAT R: ATTCCTACAACTCAATCTTACCTA | 60°C y 168pb |
F: Forward; R: Reverse.
Tras la MSP se procedió a la carga del volumen total de PCR en un gel de agarosa al 3%, con bromuro de etidio a una concentración 10μg/mL, para comprobar la correcta amplificación del ADN producto de PCR y el estado de metilación. El ADN se visualizó y se obtuvo una imagen digital del gel empleando el programa QuantityOne 4.6.1.
Cultivos celulares de H1299Como control positivo de expresión se utilizaron células H1299, de carcinoma de pulmón de células no pequeñas, que fueron cultivadas en Dulbecco¿s Modified Eagle¿s Medium de Gibco (San Diego, CA, EE. UU.) suplementado con suero bovino fetal al 10%+penicilina (100 U/mL) y estreptomicina (100mg/mL). Tras dejarlas crecer durante 48 horas, estando a una confluencia mayor del 90%, se procedió a la extracción del ADN del cultivo con el FlexiGene DNA Kit (Qiagen, Alemania) siguiendo las instrucciones del fabricante, tras despegarlas de los frascos de cultivo con tripsina de Gibco (San Diego, CA, EE. UU.). Todos los experimentos se realizaron por duplicado en 3 ensayos diferentes para garantizar la fiabilidad y reproducibilidad del ensayo.
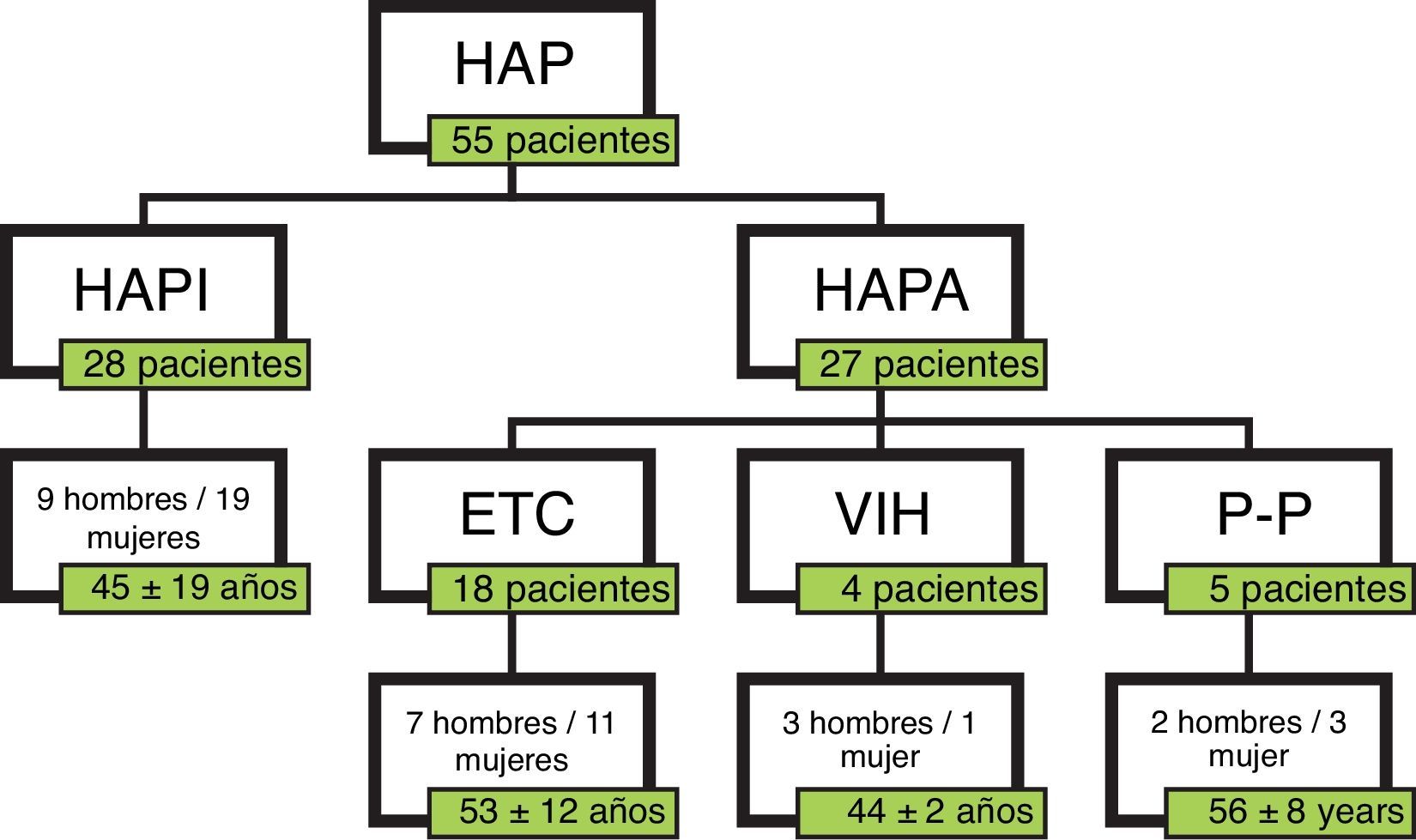
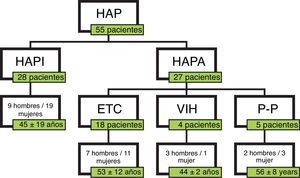
ResultadosPacientesSe incluyeron 55 pacientes, 28 diagnosticados de HAPI y 27 de HAPA. En el momento del diagnóstico 7 pacientes se encontraban en clase funcional I, 19 pacientes en CF II, 25 en CF III y 4 en CF IV (fig. 1). En total, 34 pacientes fueron considerados buenos respondedores. Se consideró buena respuesta al tratamiento tras 6 meses de mejoría de al menos una CF, disminución del propéptido natriurético cerebral (proBNP) e incremento de la distancia recorrida en el TM6M un mínimo del 15%. Las características de los pacientes se muestran en la tabla 2.
Características de los pacientes en función del tipo de HAP. ETC: enfermedad del tejido conectivo; HAP: hipertensión arterial pulmonar; HAPA: hipertensión arterial pulmonar asociada; HAPI: hipertensión arterial pulmonar idiopática; P-P: hipertensión porto-pulmonar; VIH: virus de la inmunodeficiencia humana.
Características clínicas y hemodinámicas de los pacientes
| Características | Valores |
|---|---|
| Número | 55 |
| Género | 20M/35 F |
| Edad de diagnóstico (años) | 49±16 |
| PaPm (mmHg) | 49±14 |
| PaPs (mmHg) | 70±19 |
| RVP (mmHg.l−1.m−1) | 8.2±3.3 |
| IC (l.m−1.m−2) | 2.4±0,7 |
| TM6M (m) | 415±146 |
| Tipos de HAP | 28 HAPI/27 HAPA |
Los valores se expresan como media±desviación estándar.
F: femenino; IC: índice cardiaco; M: masculino; PaPm: presión pulmonar media; PaPs: presión pulmonar sistólica; RVP: resistencia vascular pulmonar; TM6M: test en marcha de 6 minutos.
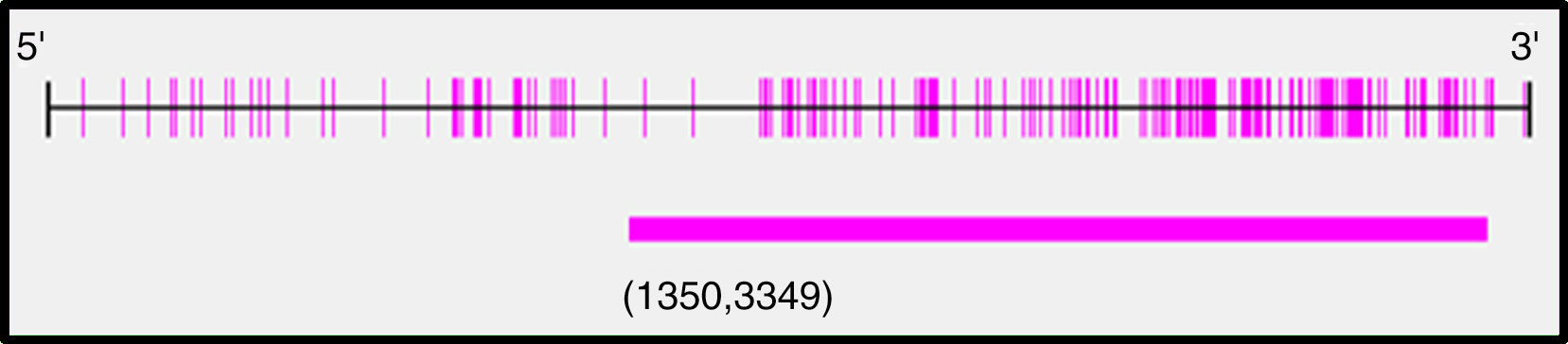
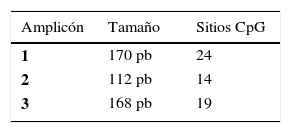
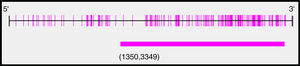
Tras el análisis de los últimos 2.000 pb de la región promotora del gen BMPR2 con el programa Methyl Primer Express® Version 1.0 se identificó una larga isla CpG, de 1.999 pb, susceptible de metilación (fig. 2). Los amplicones seleccionados para la amplificación de todo el fragmento mostraban longitudes y número de sitios CpG diferentes para cada uno de ellos, siendo el amplicón 1 el más grande y el amplicón 3 el que contaba con más sitios CpG (tabla 3).
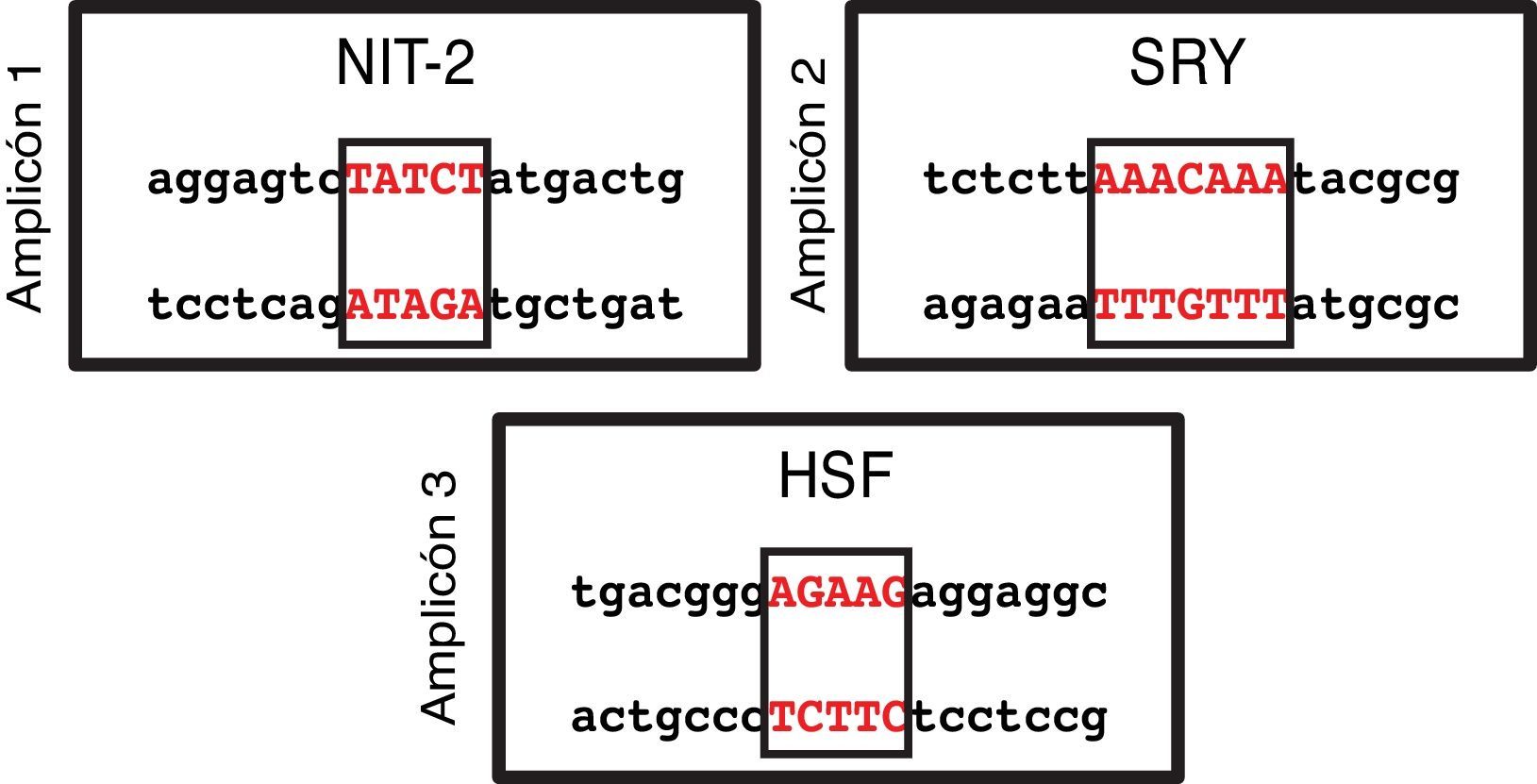
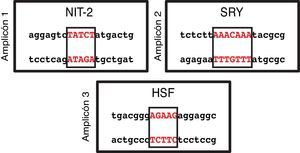
En base a la secuencia introducida en el programa informático MatInspector y la información estructural almacenada en su base de datos, se reconocieron sitios de unión a factores de transcripción en los 3 amplicones analizados. Utilizando una fiabilidad ≥ 90% cada uno de los amplicones mostró varios sitios de unión. Siendo más restricitivos y aumentando la fiabilidad al 100% solo encontramos un sitio de unión a factores de transcripción en cada uno de los amplicones (fig. 3). En el amplicón 1se localizó NIT-2 (Global-acting regulatory protein), en el amplicón 2 sex-determining region Y, y en el amplicón 3 heat shock factor. Sin embargo, ninguno de estos 3 sitios de unión a factores de transcripción detectados estaba situado entre los sitios CpG localizados.
Análisis de metilación de la región promotora del gen BMPR2Para la modificación mediante bisulfito se utilizó una concentración de ADN genómico de 450ng de cada muestra La concentración media obtenida de ADN bisulfatado fue 26,68±9,39ng/μL. La concentración más baja obtenida, en uno de los pacientes, fue de 10ng/μL y la más alta fue de 71ng/μL.
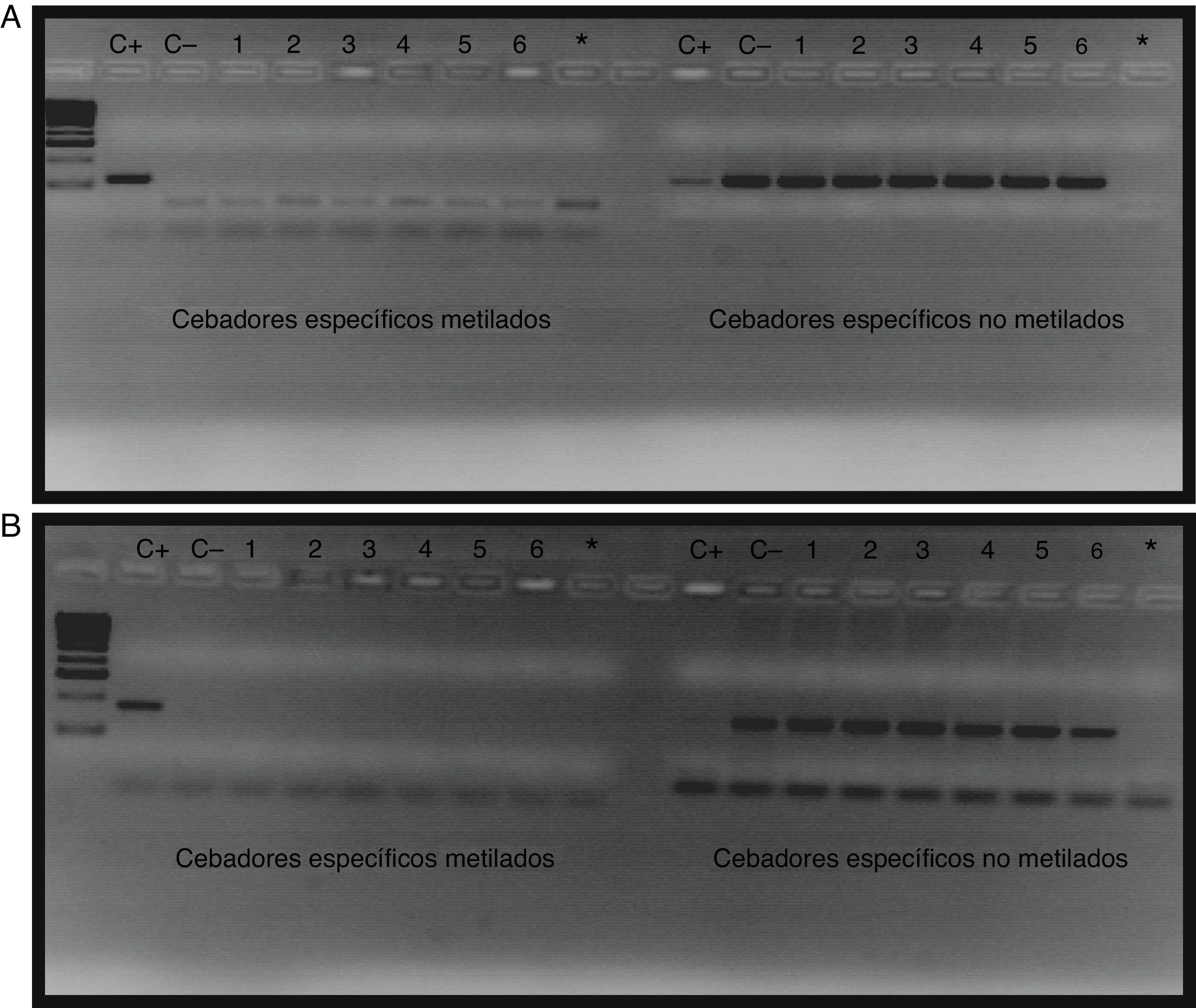
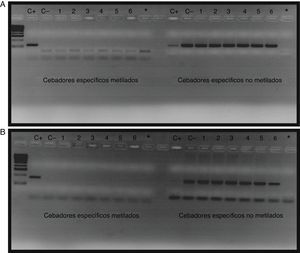
El estado de metilación de la región promotora del gen BMPR2 analizada, tanto en los 55 pacientes con HAP como en los 50 individuos control, fue negativa. Ningún paciente mostró signos de metilación tras el mapeo epigenético. En la figura 4 puede observarse un ejemplo del patrón de metilación del amplicón 3, observado en 6 pacientes escogidos al azar entre los 55 incluidos en el estudio.
Amplificación de la región promotora del gen BMPR2 en pacientes con HAP A) y controles B). Los pocillos 1-6 corresponden al resultado de la amplificación de la PCR metilada específica y a la PCR no metilada específica de los pacientes, los pocillos C+ corresponden a los controles metilados, los pocillos C− a los controles no metilados y los pocillos * a los blancos con H2O.
Las células H1299, que expresan de manera endógena el gen BMPR2, fueron utilizadas para la extracción de ADN y poder comprobar si en un tejido específico donde se expresa, la región promotora del gen a estudio se encuentra metilada. Nuestro trabajo no ha encontrado ningún indicio de la existencia de metilación en esta región para ninguno de los amplicones analizados. En la figura 5 se puede observar un ejemplo del patrón de metilación, del amplicón 2, presente en las 6 réplicas del cultivo celular H1299.
Amplificación de la región promotora del gen BMPR2 en células H1299. Los pocillos 1-6 corresponden al resultado de la amplificación de la PCR metilada específica y a la PCR no metilada específica de las réplicas de los cultivos celulares de H1299, los pocillos C+ corresponden a los controles metilados, los pocillos C− a los controles no metilados y los pocillos * a los blancos con H2O.
La epigenética es la rama de la genética que se encarga de estudiar y describir los cambios en el fenotipo o en la expresión de genes que no están involucrados en la secuencia del ADN en sí18–22. En nuestra serie de pacientes, en la región promotora analizada, no se han encontrado resultados positivos para la metilación de los sitios CpG, a pesar de ser una región que contiene un gran número de uniones a factores de transcripción y una larga isla CpG susceptible de metilación. Los mismos resultados se han obtenido para los controles estudiados, indicando que, según nuestro análisis, la metilación de la región promotora del gen BMPR2 no parece asociarse a la aparición, desarrollo o penetrancia de la HAP.
El gen BMPR2 se expresa en las células del músculo liso de las arterias pulmonares y los perfiles de metilación del genoma humano son específicos de tejido. Sin embargo, por el alto coste que supone buscar los niveles de metilación global de todo el genoma, se están empezando a utilizar perfiles específicos en tejidos de sustitución, como la sangre27,28. Para este estudio hemos utilizado el ADN de leucocitos de sangre periférica, algo muy habitual para el mapeo epigenético cuando acceder al tejido diana supone un problema27. Algunos estudios, como el de Li et al. han descrito que se puede utilizar sangre periférica como tejido para determinar los patrones de metilación en diferentes genes relacionados con distintos tipos de cáncer, dando diferencias significativas entre pacientes y controles, aunque no en todos los casos28. La ventaja de utilizar sangre periférica es que el ADN es muy fácil de obtener y de una alta calidad, lo que lo está convirtiendo en un método muy extendido. Se ha sugerido que los procesos inmunológicos asociados a la inflamación en diferentes enfermedades pueden alterar el patrón de metilación en las subpoblaciones de leucocitos28, lo cual exige un mayor conocimiento del origen y la naturaleza del ADN de leucocitos de sangre periférica para confirmar que este se puede utilizar como sustituto del ADN específico de tejido para determinar los patrones de metilación de un gen con una expresión de difícil acceso. Hamid et al.29,30 han descrito expresión del gen BMPR2 en linfocitos B humanos, confirmando la validez del diseño de nuestro análisis en sangre periférica. Otros genes que pueden provocar cambios en el fenotipo de los pacientes, como el ACVRL1 y endoglina, también se expresan, al igual que el BMPR2, en las células del músculo liso de las arterias pulmonares. Por todo ello, se buscaron patrones de metilación en el gen BMPR2 en células del músculo liso de las arterias pulmonares que sí expresen este gen, siendo negativos los resultados. Este dato podría indicarnos que, tal vez, en estado de salud la región promotora de dicho gen se encuentre desmetilada y que si se metilase podría alterar la expresión del gen o incluso silenciarlo. Sería necesario, para confirmar el patrón de metilación en pacientes con HAP, repetir el estudio utilizando el ADN específico de tejido, células del músculo liso de las arterias pulmonares o incluso utilizar modelos animales donde no resulte complicado acceder al tejido afectado.
La búsqueda en la base de datos de sitios de unión a factores de transcripción MatInspector, identificó 3 potenciales sitios de unión: NIT-2 (global-acting regulatory protein), sex-determining region Y, y Heat shock factor31–33. Estos factores de transcripción podrían alterarse si existiese un sitio CpG metilado en su secuencia, lo cual sería muy interesante puesto que estos factores de transcripción poseen importantes efectos reguladores sobre diversos promotores celulares y virales y también pueden actuar como activadores transcripcionales. Por lo tanto, si la región promotora de este gen se encontrase metilada y alterase la secuencia de algunos de estos sitios de unión, impidiendo el correcto ensamblaje de los factores de transcripción, podría ocasionar graves efectos fisiológicos provocados por la desregulación transcripcional del gen BMPR2.
Una de las características principales de los cambios epigenéticos es que son cambios heredables y tal vez, por ello, lo ideal sería analizar estos cambios en pacientes con HAPF. Por otro lado, otros mecanismos epigenéticos como la modificación de las histonas, remodelación de la cromatina o el ARN interferente podrían estar involucrados en la HAP resultando interesante su análisis ya que pueden activar o desactivar los genes que controlan el crecimiento celular, la proliferación y la apoptosis. Por el contrario, la desmetilación de sitios CpG se ha asociado con la sobreexpresión de oncogenes dentro de las células cancerígenas. Esta desmetilación puede causar una desregulación de la actividad transcripcional del gen hipometilado. Sin embargo, en nuestro estudio tampoco se han encontrado trazas de que el gen BMPR2 se encuentre en este estado, al no haber diferencias entre pacientes y controles.
La determinación del estado de metilación (metilado o no metilado) utilizado en este estudio, se ha realizado por visualización en gel de agarosa, que es una técnica observador dependiente. Se puede mejorar con el uso de una estrategia de cuantificación a través de la metilación cuantitativa en qPCR específica de metilación. Este método podría permitir la búsqueda y la estimación del porcentaje de metilación de la cohorte de pacientes para el gen estudiado, en lugar de observar simplemente presencia o ausencia de metilación34,35.
Una de las limitaciones potenciales de este estudio es el número relativamente pequeño de pacientes analizados aunque el hecho de no encontrar ningún caso parece bastante concluyente.
En conclusión, dada la naturaleza compleja y multifactorial de la HAP, parecía probable que los eventos epigenéticos pudiesen influir en el establecimiento o progresión de la misma. Aunque algunos autores han sugerido que pueden existir cambios epigenéticos en la región promotora del gen BMPR2 en pacientes con HAP, en nuestro estudio no hemos encontrado diferencias en el patrón de metilación para pacientes y población sana, a pesar de que la región a estudio probablemente es la más adecuada para este tipo de análisis, ya que contiene secuencias de unión a numerosos factores de transcripción que podrían alterar la función del gen en caso de encontrarse modificados por algún cambio epigenético.
FinanciaciónEste estudio ha sido financiado con los proyectos IN-202-05 de SOGAPAR, CO-0118-2012 de Actelion Pharmaceuticals e INBIOMED 2009-063 Xunta de Galicia.
Autoría/colaboradoresGP ha realizado el diseño del estudio, el análisis genético, el análisis de datos, las correlaciones y la redacción del manuscrito. AB se ha encargado de la recolección de muestras y datos clínicos, del análisis de datos y de la redacción del manuscrito. DV ha realizado el diseño y coordinación del estudio, el análisis de datos y la redacción del manuscrito. Todos los autores han leído y aprobado el manuscrito final.
Conflicto de interesesAdolfo Baloira ha recibido becas de investigación de Actelion y ha actuado como consultor de Actelion, GSK, Pfizer, Lilly, Bayer y Ferrer.
El resto de los autores declaran no tener conflicto de intereses.
Queremos expresar nuestro agradecimiento a los pacientes que han dado su consentimiento para participar en este estudio. También dar las gracias a los médicos que han facilitado la inclusión de pacientes (Carlos Vilariño Pombo, José Manuel Cifrián y Olalla Añón) y, finalmente, nos gustaría agradecer la colaboración de la Asociación Española de Hipertensión Pulmonar, Programa de Apoyo a las Capacidades Biomédicas (BIOCAPS) FP7-REGPOT316265.