Al igual que sucede con el resto de la patología, el estudio del asma se ha venido realizando tradicionalmente desde los postulados marcados por la ciencia reduccionista. Ese modelo sigue aportando respuestas a las preguntas teóricas y prácticas que las enfermedades plantean pero no nos ofrece una visión completa de su complejidad y multidimensionalidad. Para superar esta limitación surge la medicina orientada hacia sistemas basada en la aplicación de los conceptos y herramientas de la biología de sistemas. La biología de sistemas es una estrategia analítica transdisciplinar que, a partir de los datos generados por las ciencias ómicas, permite relacionar los elementos de un organismo o sistema biológico, comprender las propiedades emergentes del mismo y generar modelos matemáticos capaces de predecir su comportamiento dinámico. La aplicación de la biología de sistemas al asma comienza a dar ya los primeros pasos. Hoy el reto principal es comprender la necesidad del cambio de enfoque. El punto de partida pasa por abandonar la idea del asma como enfermedad exclusiva de la vía aérea considerando que en su patogénia participa todo el pulmón y, aún más, que posiblemente se trate, al menos en parte, de un proceso sistémico. Vistas nuestras limitaciones actuales, entender el asma y diseñar estrategias terapéuticas personalizadas para cada paciente, exige pensar en medicina de sistemas.
As happens with the rest of pathology, the study of asthma has been traditionally conducted from postulates set by reductionist science. That model still provides answers to theoretical and practical questions that establish diseases, but does not offer us a complete view of their complexity and multidimensionality. To overcome this limitation has emerged medicine directed towards systems based on the application of biological systems concepts and tools. Biological systems is a cross-disciplinary strategy which, from the data generated by the “-omic” sciences, helps to relate the elements of an organism or biological system, to understand the properties arising from the same and to generate mathematical models capable of predicting their dynamic behaviour. The application of biological systems to asthma starts is starting to make ground. The main challenge today is to understand the need to change focus. The starting point is to abandon the idea that asthma is exclusively an airways disease and considering that the whole lung is involved and, even more, the possibility that it is, at least in part, a systemic process. In view of our current limitations, to understand asthma and design personalised treatment strategies for each patient, requires thinking of systems medicine.
Más allá de su naturaleza inflamatoria, si algo destaca en el campo del asma como una verdad incuestionable, es la complejidad y multidimensionalidad. Ese comportamiento plural queda puesto de manifiesto a distintos niveles. Se trata de una enfermedad poligénica, con un patrón de herencia que no sigue los modelos mendelianos sencillos1–3, y donde intervienen un buen número de células y mediadores, cuya importancia varía durante la historia natural4–6. Asimismo, la expresión clínica puede adoptar fenotipos diferentes, no siempre estáticos y por tanto cambiantes con el paso del tiempo7–9, siendo frecuente la coexistencia adicional de comorbilidades que modifican, influyen o alteran la evolución, la respuesta terapéutica y el pronóstico a corto, medio o largo plazo10,11. Aún más: diversos trabajos han demostrado hasta qué punto, en esta entidad, la concomitancia de estímulos inflamatorios agregados, pulmonares o extrapulmonares, agrava la propia evolución del cuadro respiratorio12,13.
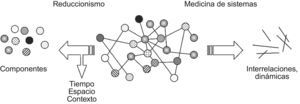
Frente a todo ello, y de acuerdo con el método seguido por la ciencia desde el siglo XVII, la orientación tradicional del asma ha mantenido un planteamiento centrado en la linealidad causa efecto y asentado sobre el postulado reduccionista de fragmentar el objeto a estudio en partes más simples, esperando que dicha segmentación permita dilucidar el funcionamiento del conjunto14. Bajo tales premisas, hemos podido sin duda escrutar y solucionar muchas de las cuestiones teóricas y prácticas que la enfermedad plantea. La mejor prueba es que su tasa de mortalidad o el número de ingresos hospitalarios han experimentado en las últimas décadas unas reducciones notables15,16. No obstante, y a pesar de los éxitos obtenidos, comienza a crecer la idea de que esta orientación muestra señales de agotamiento, al dejar de explicar la ocurrencia de situaciones de dinámica no lineal y no aportar una visión holística del tema14. De hecho, ni curamos el asma, ni conseguimos siempre el control adecuado de la enfermedad, ni tampoco parece que estemos desarrollando alternativas terapéuticas con ventajas competitivas respecto a las disponibles14, problema por otro lado también presente en otros muchos campos de la patología17.
Si aceptamos la realidad de lo expuesto, deberíamos convenir que quizás haya llegado el momento de establecer nuevas estrategias capaces de integrar toda la información conocida respecto a los elementos que conforman la realidad llamada asma y disponer así de una cartografía detallada de su complejidad. A esa aproximación se le conoce bajo el nombre genérico de medicina orientada hacia sistemas, y la herramienta metodología a aplicar biología de sistemas18–21.
Medicina orientada hacia sistemasEn contraposición al planteamiento reduccionista convencional del «divide y vencerás», la medicina orientada hacia sistemas pretende identificar no tanto las piezas constituyentes del problema, que también, sino, sobre todo, la naturaleza, dirección y características de las relaciones que existen entre aquellas20,22. El principio subyacente es que solo de ese modo será factible capturar las cualidades emergentes del conjunto (propiedades no justificables por la simple adición de las partes) y entender finalmente el comportamiento dinámico global22. La vida misma representa un paradigma claro de propiedad emergente. No brota del DNA, del RNA, de las proteínas, de los carbohidratos o de los lípidos. La vida surge de sus acciones e interacciones.
Este cambio de enfoque (de arriba a abajo, de lo general a lo particular) pretende superar los errores que se generan al intentar comprender fenómenos a escalas superiores partiendo de escalas inferiores, olvidando que, en lo complejo, el todo es más que la suma de las partes o, quizás mejor, que el todo es distinto a la suma de las partes («los árboles impiden ver el bosque»)19,22,23. En palabras de Denis Noble, la innovación esencial exige descartar por erróneo el modelo imperante de dirección única en la cadena de causas y efectos (desde los genes hasta el organismo) y complementarlo con la causación en sentido descendente, asumiendo, entre otros, algunos principios fundamentales bien demostrados en los sistemas biológicos, donde: a) la funcionalidad es multinivel; b) la información no se produce de forma unidireccional, c) la transmisión de la herencia no descansa solo en el ADN; y d) no existen niveles privilegiados de causalidad24.
La medicina orientada hacia sistemas simboliza la traducción y aplicación al campo médico de la teoría de los sistemas complejos no lineales que interactúan con el medio. Esos sistemas, ubicuos en nuestro entorno (desde reacciones químicas autocatalíticas hasta procesos de índole social y cultural), se caracterizan básicamente por los siguientes extremos:
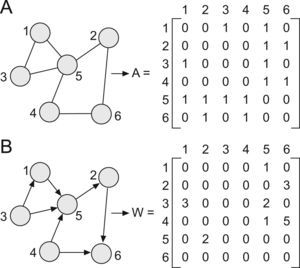
Primero. La amalgama de variables que los constituyen (aquí genes, moléculas, células…) despliegan un patrón de conectividad heterogéneo (libre de escala), con pocos elementos altamente enlazados (concentradores o hubs) y muchos con pocos enlaces25,26. A partir de un determinado grado de abstracción, esos componentes pueden ser traducidos a una serie de nodos o vértices relacionados entre sí por conexiones o aristas. Los nodos y las conexiones forman una red o, en lenguaje matemático más formal, un grafo (figs. 1 y 2)26. Los sistemas biológicos complejos adoptan muchas veces un diseño modular al agruparse los vértices en conjuntos muy interconectados y con funciones comunes25,26.
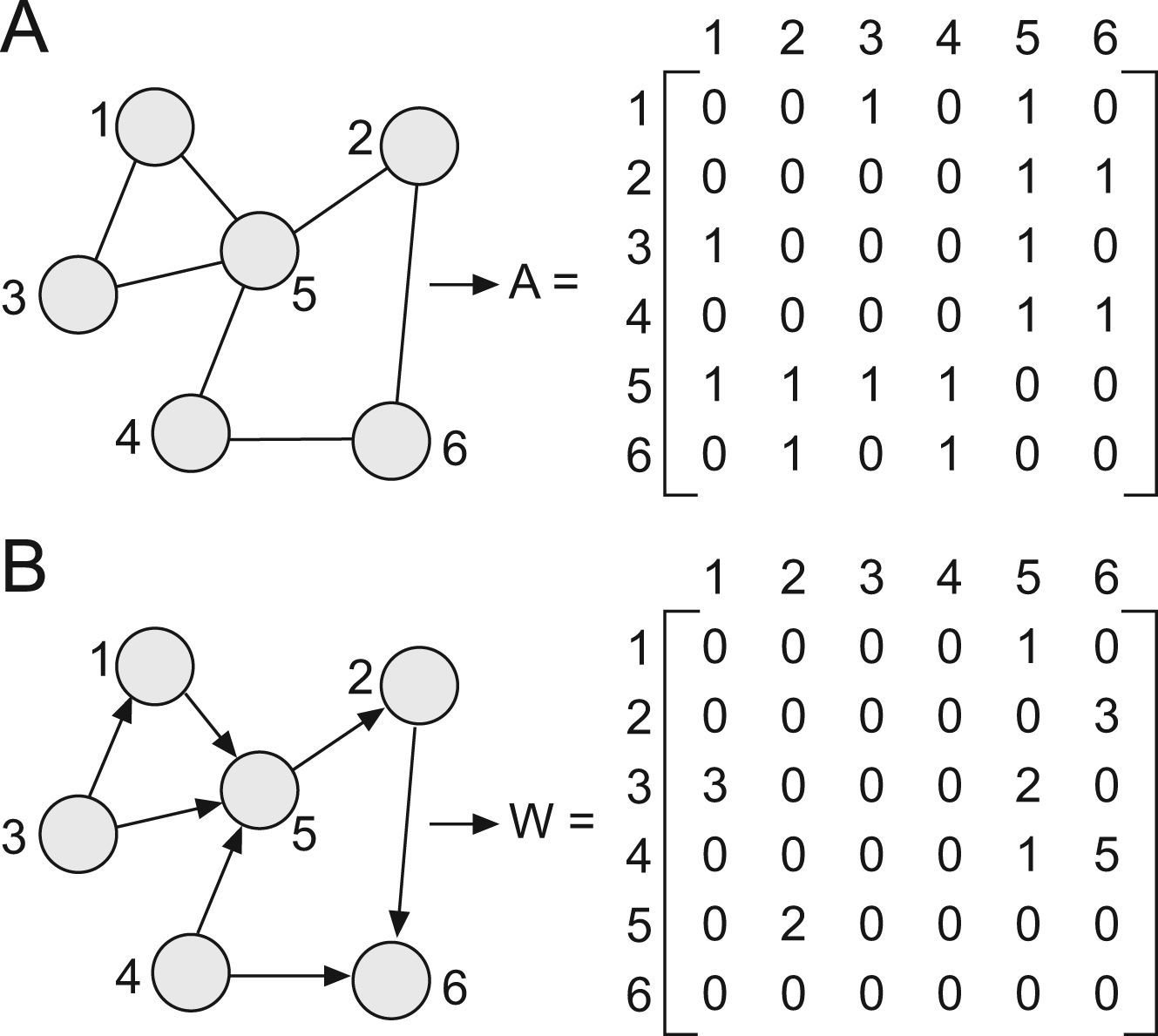
Representación en forma de grafo de una red. Un grafo G es un par G (V,E), donde V es un conjunto finito de puntos llamados vértices o nodos y E un conjunto de aristas o enlaces que conectan a los nodos de un grupo. Los enlaces pueden ser no dirigidos (simples) o dirigidos (las conexiones entre los nodos poseen un sentido). Todo grafo puede representarse mediante una matriz y el contenido de esta depende de los enlaces asociados. Existen 2 tipos de matrices: la matriz adyacente (A) y la matriz de peso (W). Cada elemento de la matriz A expresa la presencia (1) o ausencia (0) de una conexión entre nodos en una red no dirigida (fig. 2.A); nótese además que mientras que el nodo 5 tiene 4 conexiones con el resto de los elementos de la red, el número de enlaces de los demas vértices es solo de 2. La matriz W resume la fuerza de cada conexión en una red dirigida; por ejemplo, la conexión dirigida desde el 2–6 (w26=3) es más débil que la existente desde el 4–6 (w46=5) (fig. 2.B).
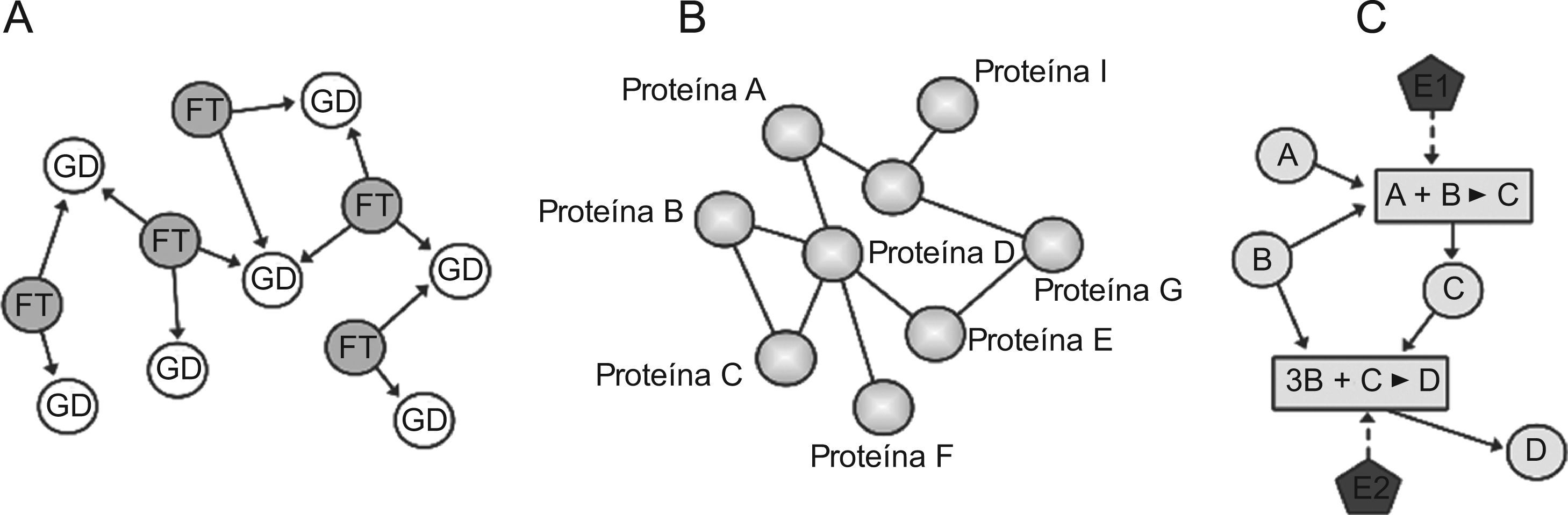
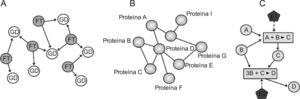
Esquema de 3 tipos de redes biológicas: A) una red reguladora transcripcional con 2 componentes (factor de transcripción y genes diana); B) una red de interacción proteina proteina (2 proteinas están conectadas si existe acoplamiento entre ellas), y C) una red metabólica construida considerando los reactantes, las reacciones químicas y los enzimas. FT: factor de transcripción; GD: genes diana; E1 y E2: enzimas 1 y 2.
Dado que hay una dinámica de interacciones fluida entre sus componentes, este tipo de sistemas pueden potencialmente existir bajo muy diferentes estados. No obstante, y a través de un proceso de «auto-organización», el sistema adopta un número reducido de configuraciones «estables» que le permiten mantener su estructura función esencial («orden emergente»)27.
Segundo. Como acabamos de avanzar, el conjunto tiende a presentar un alto grado de robustez frente a las perturbaciones, de tal manera que el fracaso temporal o permanente de algunos de sus componentes tiene a menudo un impacto escaso o nulo sobre el funcionamiento global28–30. Por definición, robustez se diferencia de estabilidad u homeostasis en un detalle capital: su objetivo es conservar la actividad del sistema más que el estado del sistema30,31. Por definición también, robustez, homeostasis y estabilidad serán conceptos equiparables cuando la función a preservar mantiene per se el estado del sistema30. Conviene además recordar que la robustez de un subsistema a menudo facilita la homeostasis del sistema a un nivel superior30.
En biología, la robustez se debe no tanto a la presencia de copias de un elemento dado con capacidad para compensar su fallo (redundancy), sino, sobre todo, a la puesta en marcha de una misma tarea a partir de elementos estructurales diferentes (degeneracy), la modularidad, la activación de mecanismos de retroalimentación y la existencia de hubs29–31. La conservación de la estabilidad de los parámetros internos no siempre suponen una ventaja para el organismo y las células cancerosas son un buen ejemplo.
Tercero. Los sistemas complejos no lineales exhiben una dependencia sensitiva respecto a las condiciones iniciales: frente a un cierto estímulo, disparidades minúsculas en la situación de partida del propio sistema darán origen a respuestas diferentes27,32. Dicho con otras palabras, aunque los factores desencadenantes tengan la misma magnitud, no necesariamente ocasionan sucesos de la misma magnitud ya que, con frecuencia, hay ausencia de proporcionalidad entre causas y efectos: causas pequeñas pueden generar efectos enormes y viceversa. Al fallar el principio de proporcionalidad, el comportamiento es de entrada poco predecible más allá de un cierto horizonte temporal. Sin embargo, el producto final, lejos de ser aleatorio y errático, contiene un orden interno subyacente regido por estrictas leyes de evolución dinámica y mensurable utilizando ecuaciones diferenciales no lineales27,32. La estimación del estado futuro del sistema será pues siempre probabilístico.
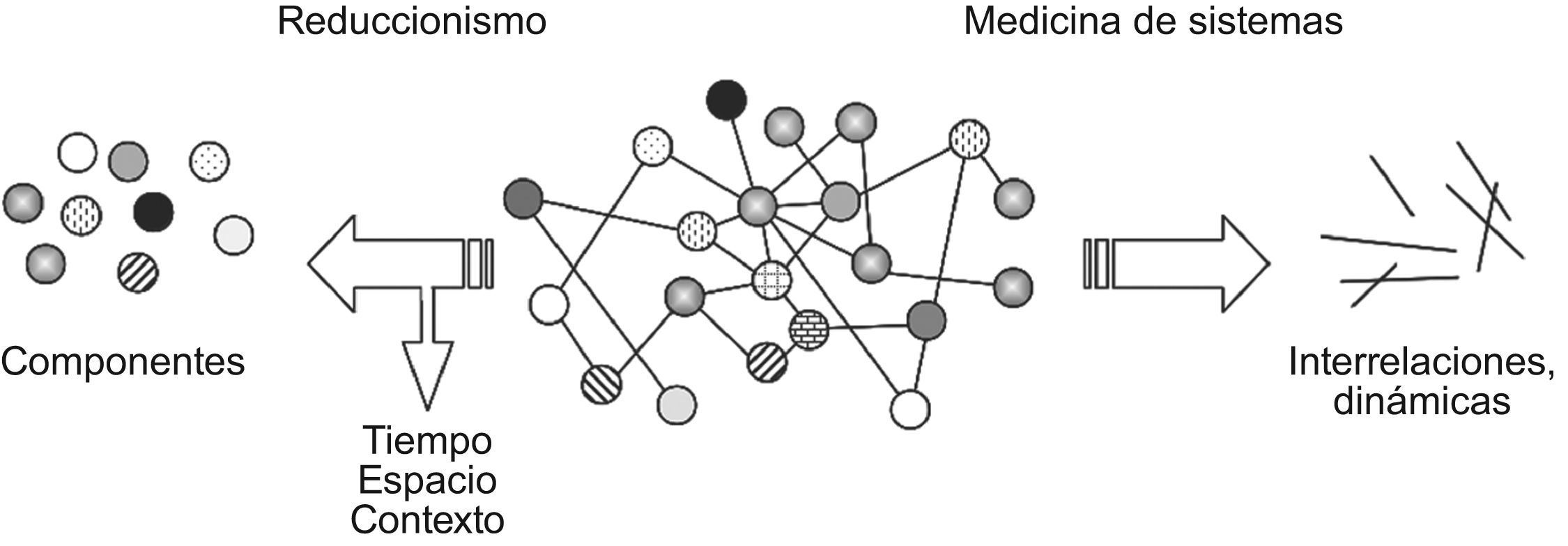
Asumir que en lo biológico el comportamiento emerge de la actividad orquestada de muchos componentes interactuando unos con otros, debe conducirnos a admitir que la condición enfermedad surge cuando acontece una desorganización dentro del sistema, suficiente para cambiar las interacciones allí existentes y comprometer parcial o totalmente las funciones y propiedades del conjunto (fallo en la robustez del sistema y pérdida de su plasticidad)20,27. Como ya se apuntó líneas arriba, lo que importa de verdad no es identificar qué piezas del puzzle no marchan, sino los links entre esas piezas y la dinámica alterada subyacente, sin olvidar los factores tiempo, espacio y contexto (fig. 3).
Reduccionismo vs, medicina de sistema. Para la medicina reduccionista, el foco de atención queda centrado prioritariamente en los componentes del problema a estudio, perdiéndose información respecto al tiempo, espacio y contexto del mismo. En la medicina orientada a sistemas, lo que se pretende es identificar no ya solo los elementos individuales, sino también sus interacciones y evolución.
Ahn et al, han resumido de un modo excelente cuáles son, en medicina, las diferencias capitales entre ontología reduccionista y ontología de sistemas, recordándonos que el inconveniente del enfoque reduccionista convencional no radica en su uso. Radica en pensar que esta es siempre la única solución (tabla 1)19,22. Parafraseando a Thomas Lemberger, la aplicación a la investigación médica, tanto básica como clínica, de la biología de sistemas abre un camino para: a) profundizar en el conocimiento de la relación genotipo fenotipo; b) aportar información relevante sobre el impacto de las interacciones entre las condiciones ambientales y el fenotipo; c) explorar nuevas perspectivas mecanísticas funcionales basadas en aproximaciones globales sin apriorismos iniciales; y d) elaborar modelos predictivos que capturen lo intrincado de los estados fisiológicos (y patológicos)20.
Medicina reduccionista y medicina orientada a sistemas: diferencias esenciales (basada en referencias 19 y 22)
| Características | Reduccionista | Orientada a sistemas |
| Principio | El comportamiento del sistema biólogico puede ser explicado por las propiedades de sus componentes | Los sistemas biológicos presentan propiedades emergentes que no se explican por la simple adición de sus componentes |
| Aproximación | Se consideran uno o pocos factores para interpretar el fenómeno sujeto a estudio | Muchos factores son tenidos en cuenta al mismo tiempo para valorar la dinámica del sistema |
| Caracteristicas del modelo | Lineal, predecible, frecuentemente determinista | No lineal, sensible a las condiciones iniciales, estocástico (probabilístico), caótico |
| Prototipo de enfermedad a aplicar | Enfermedades simples, agudas | Enfermedades complejas, crónicas |
| Ejemplos de enfermedades | Infección urinaria, apendicitis | Asma, EPOC, diabetes, enfermedad coronaria |
| Limitaciones teóricas | Resta importancia a las interacciones componente-componente y a su dinámica | Aumento de los costes y mayor consumo de tiempo |
La biología de sistemas es un campo de investigación que se preocupa del estudio comprehensivo de procesos biológicos, analizando la manera en la que todos sus componentes interactúan funcionalmente a lo largo del tiempo18,21,33. De institucionalización académica reciente, la biología de sistemas nace en la «era postgenómica» gracias a la coincidencia de 2 circunstancias: a) el desarrollo de tecnologías automatizadas de alto rendimiento que permiten la obtención de datos cuantitativos de elevada precisión; y b) el diseño de software capaz de manejar e interpretar convenientemente la información así generada34,35. La biología de sistemas constituye, en definitiva, un área de conocimiento transdisciplinar (el prefijo trans indica simultáneamente entre, a través y más allá de las fronteras disciplinares), donde convergen científicos de formación teórica muy dispar (biólogos, fisiólogos, bioquímicos, matemáticos, físicos, informáticos…) con un propósito final: integrar los experimentos de laboratorio, llamados «húmedos», con los de in silico, denominados «secos»35.
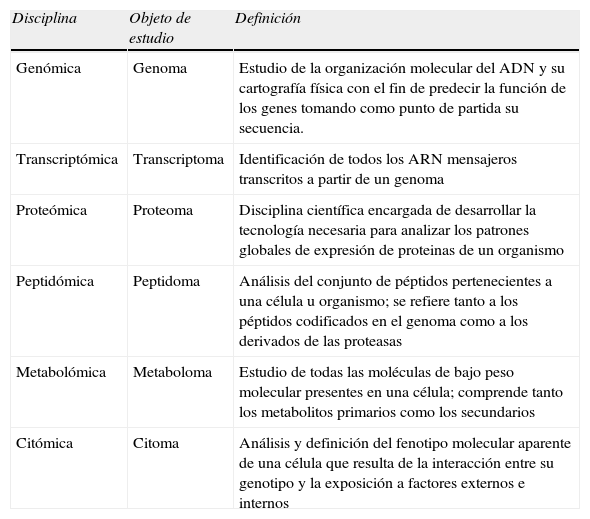
Los «húmedos» suponen la obtención y acumulación de datos obtenidos por las ciencias que estudian los genes, sus productos iniciales (transcritos de ARN) y finales (proteínas), y los productos participantes o derivados (metabolitos) de los procesos metabólicos en los que intervienen las proteínas (ciencias «ómicas»)36,37. Las tablas 2 y 3 recogen, respectivamente, las materias de interés de las principales «ómicas» y la descripción somera de algunos de los análisis y técnicas empleados en ellas.
Algunas de las principales «ómicas»: objeto de estudio y definición
| Disciplina | Objeto de estudio | Definición |
| Genómica | Genoma | Estudio de la organización molecular del ADN y su cartografía física con el fin de predecir la función de los genes tomando como punto de partida su secuencia. |
| Transcriptómica | Transcriptoma | Identificación de todos los ARN mensajeros transcritos a partir de un genoma |
| Proteómica | Proteoma | Disciplina científica encargada de desarrollar la tecnología necesaria para analizar los patrones globales de expresión de proteinas de un organismo |
| Peptidómica | Peptidoma | Análisis del conjunto de péptidos pertenecientes a una célula u organismo; se refiere tanto a los péptidos codificados en el genoma como a los derivados de las proteasas |
| Metabolómica | Metaboloma | Estudio de todas las moléculas de bajo peso molecular presentes en una célula; comprende tanto los metabolitos primarios como los secundarios |
| Citómica | Citoma | Análisis y definición del fenotipo molecular aparente de una célula que resulta de la interacción entre su genotipo y la exposición a factores externos e internos |
Descripción de algunos de los análisis y técnicas experimentales empleados en biología de sistemas
| Análisis | Técnicas |
| Análisis de la secuencia genética: secuenciación del genoma del organismo, descripción de los genes que regulan o forman parte de una red de interacción; variación genética responsable de la diferente expresión de proteínas (polimorfismos) | Secuenciación de ADN |
| Genotipado | |
| Identificación de deleciones genéticas | |
| Knockouts ARNi | |
| Análisis de la expresión génica: descripción de genes regulados positiva y negativamente en respuesta a perturbaciones genéticas o ambientales; identificación de los genes expresados en función del tejido y condiciones | Microarrays de AND |
| Marcaje de ADN | |
| Análisis de interacciones ADN-proteínas: descripción de los genes regulados por determinados factores de transcripción en condiciones definidas | Inmunoprecipitación de cromatina |
| Biochips (localización de sitios de unión, Chips-chips) | |
| Análisis de interacciones proteína-proteína: descripción de las proteínas presentes en complejos enzimáticos, poros nucleares, citoeesqueleto; identificación de proteínas que modifican otras proteínas durante las cascadas de señalización | Sistema doble híbrido |
| Purificación por afinidad | |
| Espectrometría de masas | |
| Proteómica cuantitativa | |
| Análisis de la localización subcelular proteica: localización de la síntesis de proteínas y destino de las mismas dentro de la célula | Separación celular por cell sorting |
| Marcaje molecular por medio de genes trazadores o anticuerpos |
Por su parte, los experimentos «secos» o in silico (un expresión que significa «hecho por computador o vía simulación computacional») necesitan programas informáticos que, a partir de la información surgida desde las «ómicas», puedan establecer modelos predictivos de los sistemas biológicos38,39. Las herramientas empleadas se basan en el desarrollo de algoritmos computacionales, la aplicación de modelos matemáticos (estadísticos, cinéticos, redes neuronales, modelos Markov…) y la reproducción y simulación en el ordenador del comportamiento del conjunto35,40.
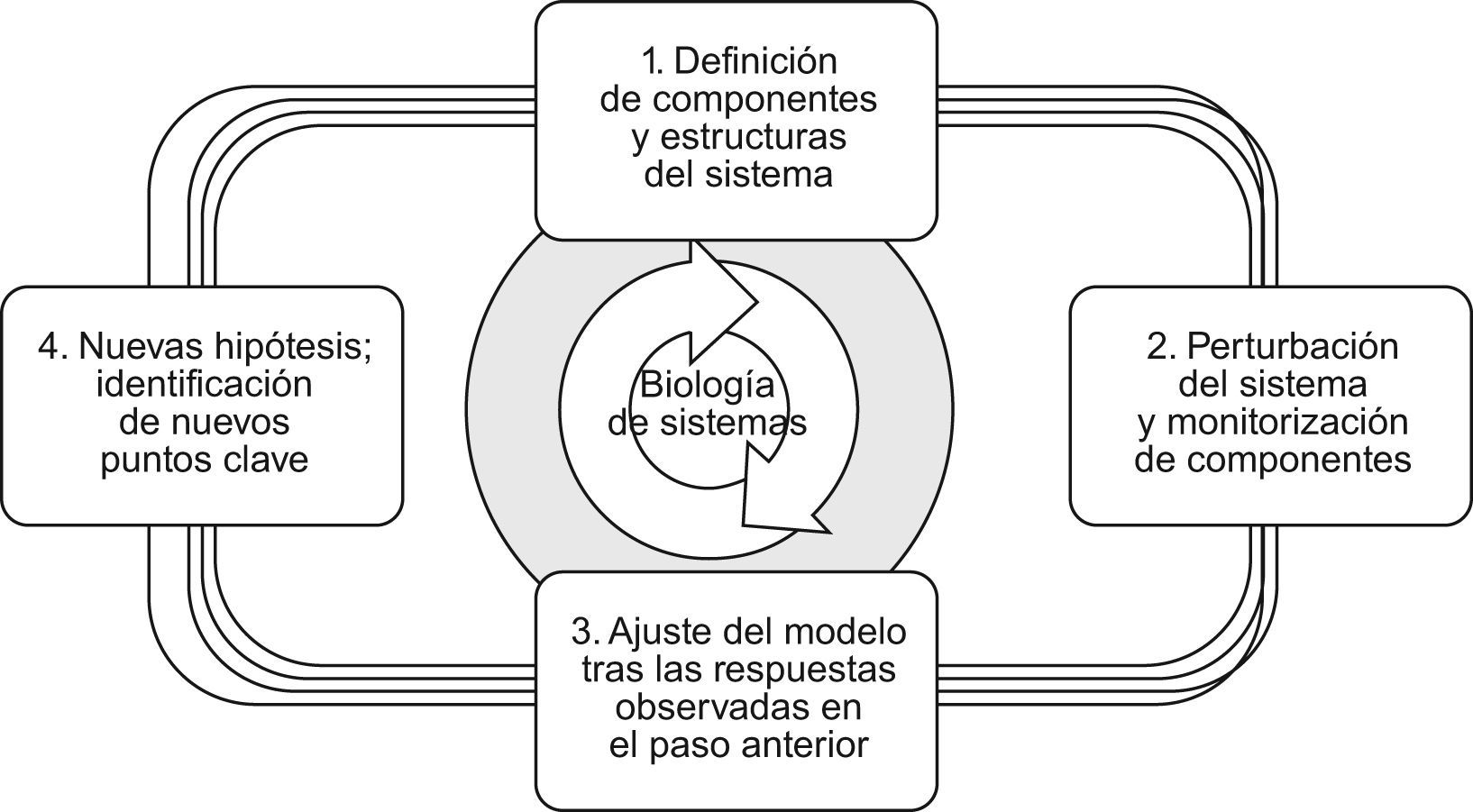
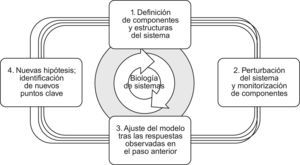
La biología de sistemas utiliza una estrategia circular en donde, de acuerdo con Kitano, cabe distinguir 4 etapas consecutivas: a) la definición de los componentes, estructura e interacciones del sistema; b) el análisis de su comportamiento frente a estímulos externos (perturbaciones) para construir el modelo inicial del mismo; c) la actualización y refinamiento del modelo a partir de las respuestas observadas en la etapa anterior y d) la formulación de nuevas hipótesis e identificación de nuevos puntos clave (fig. 4)18.
En este campo del conocimiento, el camino que resta por recorrer es todavía largo. Sin embargo, los logros ya obtenidos permiten aventurar que este modo de afrontar los problemas aportará soluciones de gran interés para desentrañar la complejidad de las redes biológicas, descifrar la patogénia de las enfermedades, identificar biomarcadores potentes, diseñar soluciones terapéuticas distintas y avanzar hacia una medicina personalizada17,19,21,35.
Medicina de sistemas en asma. Los principios para un cambio de estrategiaLa lógica e instrumentos propios de la biología de sistemas ha comenzado a aplicarse en diferentes campos de la patología. Prueba de ello son las publicaciones aparecidas al respecto sobre disfunción orgánica múltiple27, trastornos metabólicos41, cáncer42, fibrosis pulmonar43, inflamación44, tuberculosis45, autismo46, Alzheimer47, enfermedad pulmonar obstructiva crónica48, insuficiencia cardiaca49 o respuesta inmune50. En el campo del asma también estamos asistiendo ahora a la aparición de los primeros estudios51–54 que nos están haciendo avanzar en la compresión del asma al desentrañar una parte del puzzle no sospechada con anterioridad.
Pero, a mi modo de ver, y con independencia de la (r)evolución iniciada por los investigadores básicos, el problema aquí radica en que la traslación al mundo de la clínica de los nuevos datos relacionales que se vayan obteniendo será tanto mas difícil cuanto menos entendamos los médicos la necesidad de un cambio de modelo/enfoque. El punto de partida pasaría por abandonar la noción del ama como enfermedad exclusiva de la vía aérea y considerar que en su patogenia esta implicado todo el pulmón o aún más: que se trata, al menos en parte, de una patología sistémica. La idea no es nueva13,14,55. El giro copernicano radica en aceptar que si queremos una visión del asma elegante, unificadora y real (donde lo real es relacional), debemos dejar de mirar el asma a través del ojo de la cerradura, desechar su parcelamiento conceptual y convenir que, si bien el pensamiento complejo no soluciona per se los problemas complejos, ayuda a diseñar las estrategias para resolverlos.
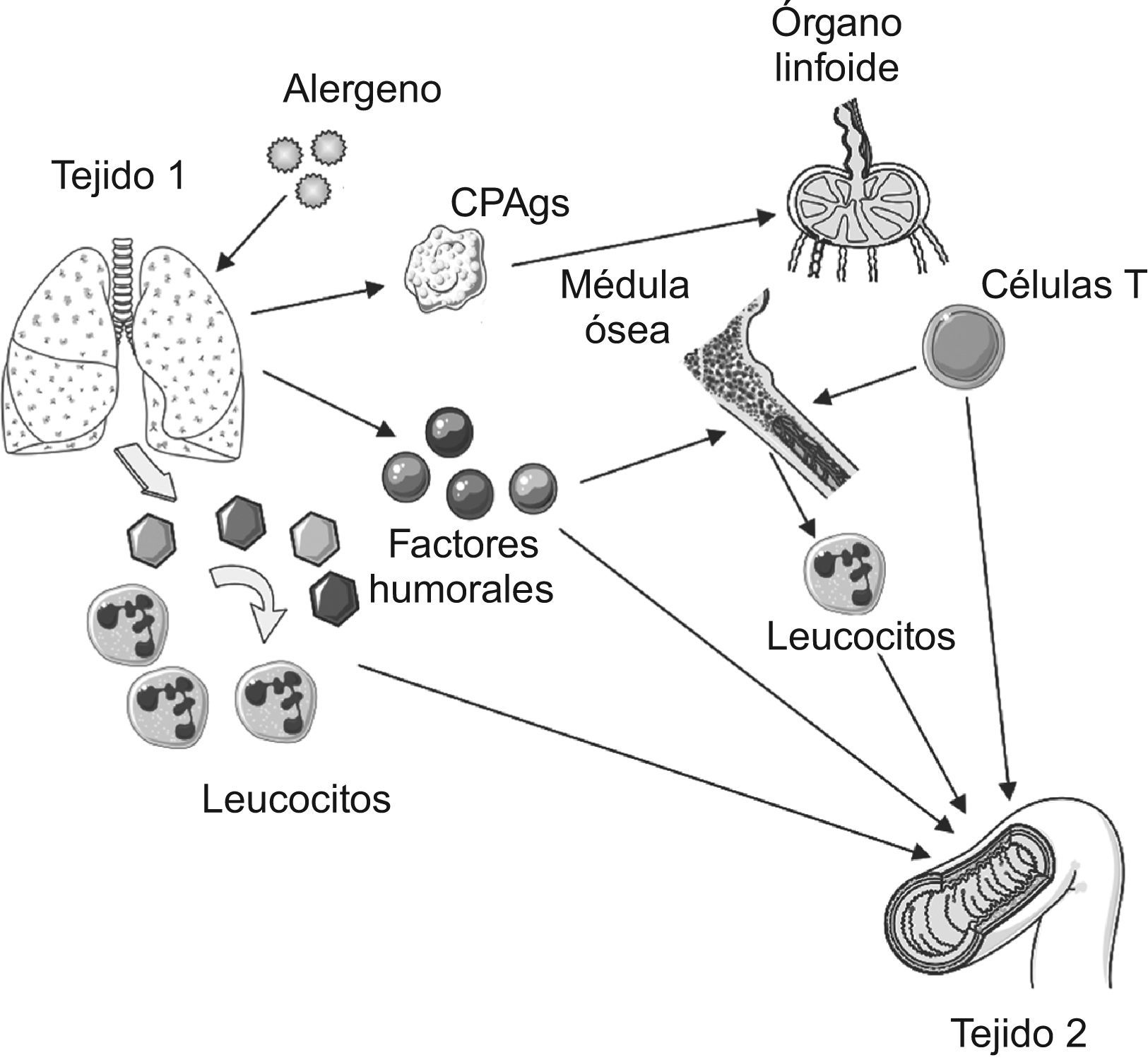
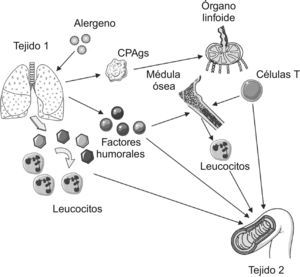
Esas son precisamente las líneas maestras del artículo de opinión, firmado por Voelkel y Spiegel, con el título: Why is effective treatment of asthma so difficult? An integrated systems biology hypothesis of asthma56. Desde su punto de vista, la explicación de la patogénia del asma quedará siempre incompleta mientras la focalicemos en las acciones, sobre el tracto respiratorio, de los eosinófilos, los mastocitos o los leucotrienos (y cito solo alguno de los paradigmas clásicos). Lo nuclear reside en integrar que el asma es, a la luz de la información actual, un proceso más general, donde mediadores inflamatorios sistémicos actúan como piezas responsables de: a) la perpetuación y expansión de las alteraciones a nivel pulmonar; b) la aparición de efectos extrapulmonares, y c) el «diálogo» e interacción entre ambos fenómenos, los locales y los generales56. En la propagación y diseminación de las señales necesarias participarían la médula ósea y el sistema nervioso14,56–58. El papel de la médula ósea ha recibido en los últimos años una atención especial al comprobarse que la inflamación pulmonar inducida por estímulos diferentes (incluyendo alergenos) conlleva la síntesis de mediadores capaces de provocar en médula la producción de células progenitoras hematopoyéticas y mesenquimales implicadas en la regulación de la inflamación pulmonar y quizás también en su expansión a otros territorios14. Togias ya apuntó esta alternativa centrando la cuestión en los efectos «generales» de los procesos alérgicos «locales»58. Basada en sus comentarios, la figura 5 sintetiza las diferentes rutas por las que la inflamación asmática podría originar una respuesta inflamatoria a distancia (extrapulmonar).
Posibles vías implicadas en el desarrollo de inflamación a distancia (tejido 2) tras el inicio de una respuesta inflamatoria, en el ejemplo por alergenos, localizada en tejido 1 (pulmón). Como resultado de la reacción alérgica en el tejido 1, tendría lugar un aumento de la expresión de las moléculas de adhesión en las vénulas postcapilares. Esta circunstancia determinaría la activación de leucocitos circulantes que alcanzarían a otros tejidos (tejido 2). Además de ello, factores humorales derivados de la reacción original migrarían a través del torrente circulatorio hasta localizarse también en el tejido 2. Paralelamente, las células presentadoras de antígenos (CPAgs) transportarían alergenos a los órganos linfoides donde los expondrán frente a los linfocitos T. Una vez activados, los linfocitos T entrarán en la circulación sanguínea y se dirigirán al lugar original de la reacción (tejido 1) o a otros territorios (tejido 2). Finalmente, las células precursoras de los leucocitos existentes en médula ósea pueden resultar activadas (por la acción de los factores humorales o de las citocinas liberadas a partir de los linfocitos) y comenzar a circular.
Pero aún en el caso de que lo sistémico fuera un epifenómeno, lo que no cabe olvidar es la complejidad intrínseca de la propia inflamación alteración disfunción pulmonar de esta enfermedad y sus cambios con el paso del tiempo. En el asma están implicadas más de 50 citocinas actuando a través de más de 20 tipos de receptores56. ¿De verdad podemos creer que su patogenia quedará al descubierto estudiando solo alguno de esos elementos?
La medicina de sistemas está aún en sus inicios y queda todavía un enorme trecho para que su metodología de investigación, la biología de sistemas, nos de las soluciones que estamos tanto tiempo buscando. El asma, y lo señalamos en la introducción, es una enfermedad compleja y entender los problemas complejos exige aplicar herramientas de estudio que superen las limitaciones de la ciencia mecanicista. Ahora ya tenemos la tecnología. Lo que necesitamos es su generalización y, sobre todo, entender el cambio de paradigma que se ha producido en el estudio de los fenómenos biológicos. Las perspectivas de la simplicidad nacieron del enfoque analítico que reduce el mundo a la unidad y que conciben la diversidad como mera combinatoria de unidades. Desde esa mirada a lo sumo puede llegar a pensarse en términos de complicación. Sin embargo, la complejidad es algo muy diferente de la mera complicación. Implica forjar un marco conceptual completamente distinto que permita concebir sistemas multidimensionales nacidos en una dinámica relacional. Vistas nuestras limitaciones actuales, comprender el asma y diseñar estrategias terapéuticas útiles para cada paciente, exige pensar en medicina de sistemas.
El contenido del presente artículo está basado en la conferencia del mismo título pronunciada por el autor durante el XXXV Simposio de Neumología (octubre 2009).