El análisis molecular del gen que codifica la alfa-1 antitripsina (AAT; gen SERPINA1) es la aproximación de referencia para la identificación de las variantes alélicas1. Entre los diferentes métodos moleculares que pueden emplearse para este fin se encuentran las denominadas sondas de hibridación o HybProbes2, las cuales permiten realizar un seguimiento de la PCR a tiempo real y, una vez terminado el proceso de amplificación, obtener información sobre las variantes genéticas presentes en una determinada región dentro del amplicón. Se trata de un ensayo de genotipado homogéneo, es decir, todo el proceso ocurre en un solo tubo sin manipulaciones adicionales entre el inicio del ensayo y la observación de los resultados. Sin embargo, aunque es una técnica muy segura, en ocasiones podemos encontrarnos con errores sobre todo en la interpretación de los resultados3.
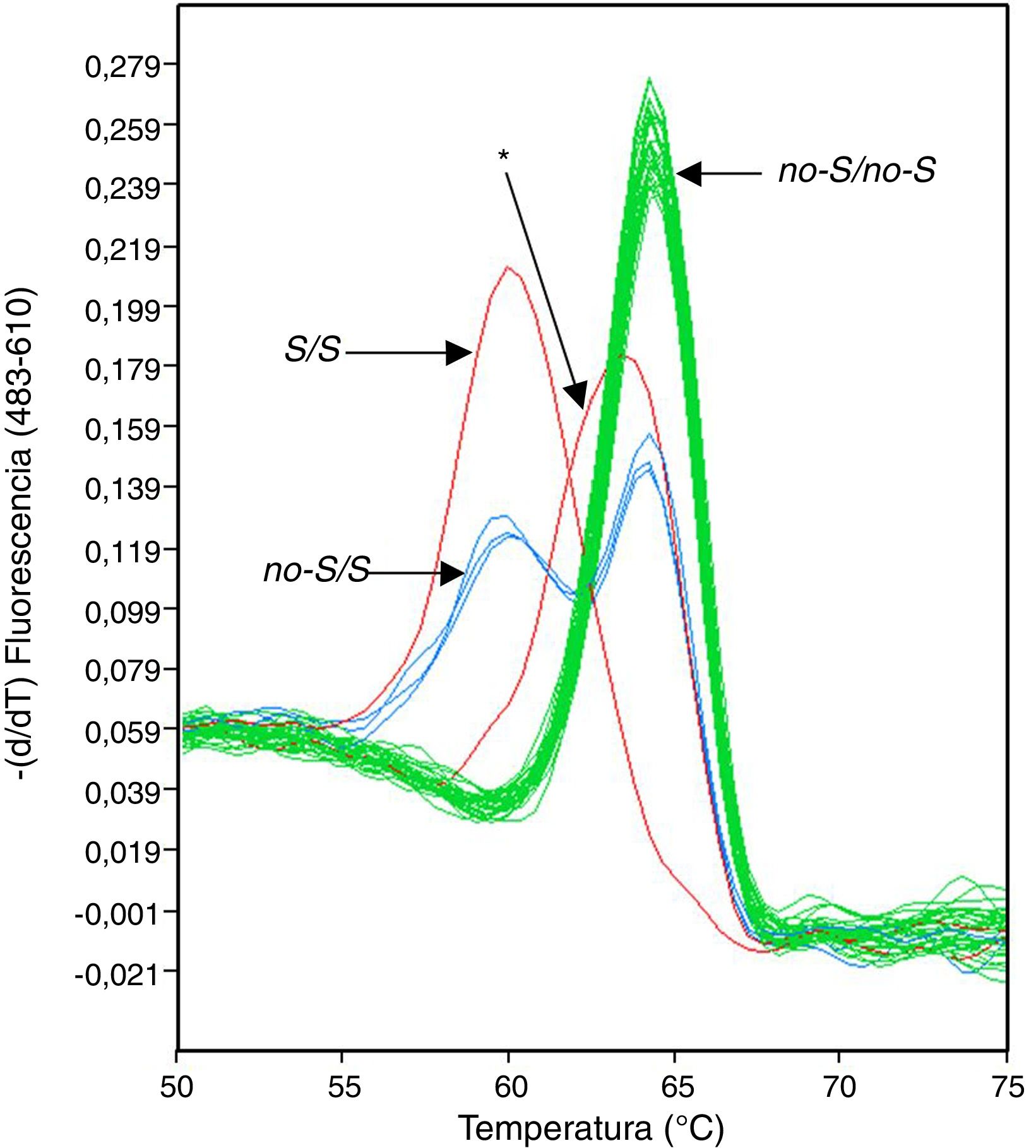
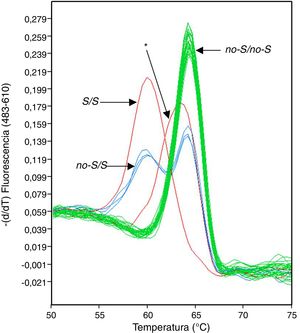
En el transcurso de un análisis centrado en la prevalencia de las variantes no-S/S y no-Z/Z del gen SERPINA1 en una población clínica de La Palma (Islas Canarias, España), con una muestra de 1.510 pacientes reclutados para este estudio independientemente del motivo que les llevó a la consulta de neumología, se detectaron 7 sujetos en los que las sondas HybProbe diseñadas para identificar las variantes no-S/S producían un pico de fusión desplazado con respecto a los picos normalmente registrados (fig. 1). En estos 7 pacientes se habían diagnosticado diversas patologías respiratorias, como enfermedad pulmonar intersticial difusa, síndrome de apnea-hipopnea del sueño o enfermedad pulmonar obstructiva crónica.
Para descartar un error en el proceso de genotipado debido a diferencias en la concentración salina de las 7 muestras de ADN afectadas, estas se volvieron a preparar y analizar, pero el software de genotipado de la plataforma de PCR a tiempo real (LightCycler 480) seguía asignando estas muestras a un grupo de genotipo diferente a los definidos por los estándares, utilizando los valores umbrales de similitud y resolución por defecto de la aplicación informática. El examen de las temperaturas de fusión indicaba una clara diferenciación entre los picos de fusión de las 7 muestras (media=63,21°C; DE=0,05) y los picos de fusión más cercanos correspondientes a muestras con genotipo no-S/no-S (media=64,18°C; DE=0,26; n=71).
Estos resultados sugerían que el gen SERPINA1 en los individuos implicados presentaba alguna mutación diferente a la S (c.863A>T; p.Glu264Val) en la región cubierta por las sondas de genotipado. Una candidata es la mutación c.839A>T (p.Asp256Val), una variante deficitaria grave con implicaciones clínicas que define, dependiendo del fondo genético, a los alelos PI*Plowell, PI*Pduarte y PI*Ybarcelona4-6. Esta posibilidad quedó descartada cuando se comprobó que el nivel sérico de AAT en los 7 pacientes se encontraba en el intervalo 93,5-167mg/dl. Aun no habiendo indicios de deficiencia grave en AAT en estos pacientes, decidimos seguir indagando en la que parecía la variante genética rara más frecuente en nuestra muestra clínica. En este sentido, en 3 de los 7 pacientes se amplificó la región codificadora del gen SERPINA1 y los intrones correspondientes, en forma de dos amplicones solapantes que fueron secuenciados. Este análisis reveló que los tres pacientes portaban en heterocigosis la mutación c.840T>C (p.Asp256Asp), precisamente en la región a la que se une la sonda ancla usada para detectar las variantes no-S/S. Además, uno de estos pacientes era heterocigótico para la mutación c.774G>A (p.Lys236Lys). Estas variantes mutantes parecen no ser deficitarias, ya que los niveles de AAT medidos en los pacientes Pi*MMD566D (166 y 142mg/dl) y en el paciente Pi*SMD566D (125mg/dl) encajan perfectamente en el intervalo de referencia para individuos con genotipo Pi*MM o Pi*MS7. Esta observación es predecible, pues se trata de dos mutaciones que no cambian la secuencia de la proteína (mutaciones sinónimas), al igual que puede esperarse que no afecten al análisis del fenotipo de estos pacientes mediante isoelectroenfoque.
La mutación c.840T>C, en el fondo alélico normal M1-Val213, en combinación con la mutación c.1093G>A (p.Asp341Asn) conforman el alelo no deficitario PI*Psaintalbans4. Puesto que esta segunda mutación no se encuentra en los tres pacientes analizados, se descartó la presencia de dicho alelo en ellos. Debido a que la PCR convencional amplifica simultáneamente los dos alelos del gen SERPINA1, no se pudo saber en qué fondo alélico normal se encontraban exactamente las mutaciones detectadas en estos pacientes. Sin embargo, sí se pudo deducir qué combinación de dichos fondos alélicos presentaba cada paciente. Interesantemente, hasta la fecha la mutación c.840T>C en el fondo M1-Ala213, o la combinación de las mutaciones c.840T>C y c.774G>A en el fondo M1-Val213, solamente han sido descritas en la población subsahariana de Sudáfrica, donde alcanzan frecuencias alélicas del 10,4 y del 4,3%, respectivamente8. Aunque su confirmación necesitaría el análisis de haplotipos, estos alelos subsaharianos podrían encontrarse en la población actual de la isla de La Palma y, presumiblemente, haberse incorporado a través de esclavos africanos traídos a la Isla de La Palma desde mediados del sigloxvi9.
Los autores agradecen a Grifols International S.A. la financiación de este trabajo de investigación.