Conocer la utilidad de las técnicas moleculares para el diagnóstico de resistencias y la situación de las resistencias a fármacos de primera línea en nuestra área geográfica.
Material y métodoDesde 2004 a 2013, 1.889 cepas de Mycobacterium tuberculosis complex aisladas en Asturias, España, fueron estudiadas mediante pruebas de sensibilidad fenotípicas (directrices del Clinical and Laboratory Standards Institute) y moleculares (INNOLiPA RIF-TB©; GenotypeMDRplus©; GenotypeMDRsl©).
ResultadosMil setecientas cincuenta y nueve cepas (94,52%) eran sensibles a todos los fármacos de primera línea y 102 cepas (5,48%) presentaban alguna resistencia: 81 cepas (4,35%) a un solo fármaco, 14 (0,75%) con polirresistencia y 7 (0,37%) multirresistentes (resistencia a rifampicina e isoniacida). En total hubo 137 resistencias a fármacos: 60 a isoniacida (3,22%), 7 a rifampicina (0,37%), 9 a pirazinamida (0,48%), 11 a etambutol (0,59%) y 50 a estreptomicina (2,68%). El 75,9% de las mutaciones detectadas (63/83) se correlacionaron con resistencia; mientras que un 24,09% de las mutaciones detectadas (20/83) no implicaban resistencia, correspondiendo 16 a una mutación silente en el codón 514 del gen rpoB. Entre un 0 y un 90% de cepas, dependiendo del fármaco que se considere, eran resistentes aunque no presentaban mutaciones en los genes incluidos en los sistemas comerciales.
ConclusionesLas técnicas moleculares resultan muy útiles sobre todo por la rapidez en la obtención de resultados, aunque estos deben confirmarse con las pruebas de sensibilidad fenotípicas de referencia. La tasa de resistencias a fármacos en nuestra región es baja y los casos de multirresistencia (0,37%) son esporádicos.
To determine the utility of molecular techniques in the diagnosis of resistance and the extent of resistance to first-line drugs in our region.
Material and methodFrom 2004 to 2013, 1,889 strains of Mycobacterium tuberculosis complex isolated in Asturias, Spain, were studied using phenotypic (Clinical and Laboratory Standards Institute guidelines) and molecular (INNOLiPA RIF-TB©; GenotypeMDRplus©; GenotypeMDRsl©) sensitivity tests.
Results1,759 strains (94.52%) were sensitive to all first-line drugs, and 102 strains (5.48%) showed some resistance: 81 strains (4.35%) were resistant to 1 single drug, 14 (0.75%) were polyresistant, and 7 (0.37%) were multiresistant (resistant to rifampicin and isoniazid). In total, 137 resistances were identified: 60 to isoniazid (3.22%), 7 to rifampicin (0.37%), 9 to pyrazinamide (0.48%), 11 to ethambutol (0.59%), and 50 to streptomycin (2.68%). Of the mutations detected, 75.9% (63/83) correlated with resistance, while 24.09% of mutations detected (20/83) were not associated with resistance; 16 of these involved a silent mutation at codon 514 of the rpoB gene. Between 0 and 90% of strains, depending on the drug under consideration, were resistant even when no gene mutations were detected using marketed systems.
ConclusionsMolecular techniques are very useful, particularly for obtaining rapid results, but these must be confirmed with standard phenotypic sensitivity testing. The rate of resistance in our region is low and multi-drug resistantcases (0.37%) are sporadic.
La persistencia de la tuberculosis (TB) está asociada a varios factores entre los que destacan: los problemas socioeconómicos, el aumento de la coinfección con el VIH y el creciente aumento de la resistencia a los fármacos antituberculosos1.
La situación actual de la tuberculosis resistente en el mundo es preocupante; se estima que anualmente se producen cerca de 500.000 casos de tuberculosis multirresistente (MDR-TB), definida como resistencia al menos a isoniacida y rifampicina2, lo que representa aproximadamente el 3,5% de todos los casos nuevos de TB. Aunque con grandes diferencias geográficas, en la mayor parte de los países del mundo ya ha sido diagnosticado algún caso de tuberculosis extremadamente resistente (XDR-TB), definida como MDR-TB con resistencia añadida a alguna fluoroquinolona y, al menos, a un fármaco inyectable de segunda línea, lo cual da una idea de la magnitud del problema3.
La incidencia de TB sigue descendiendo en España; este comportamiento epidemiológico es similar al de los países de nuestro entorno4, pero la aparición de cepas MDR y XDR-TB, ponen en riesgo el control de la enfermedad, por lo que es recomendable estudiar todas las cepas circulantes.
La tasa de incidencia de TB en Asturias en 2013 fue de 14,98 casos por 100.000 habitantes5, algo superior a la media nacional que fue de 11,88 casos por 100.000 habitantes6.
El conocimiento de la situación de las resistencias en cepas de Mycobacterium tuberculosis (M. tuberculosis) en un área geográfica, permite adecuar las pautas de tratamiento a las necesidades de la población y nos indica la idoneidad de las pautas terapéuticas existentes7.
Hay diferentes metodologías para conocer el patrón de sensibilidad de los aislamientos clínicos; el método más exacto y el que menos resultados falsos proporciona es el método de las proporciones múltiples establecido por Canetti en 19638,9, por lo que es el más utilizado en todo el mundo.
La demora inherente a las pruebas de sensibilidad convencionales hace que el interés por las técnicas moleculares sea cada vez mayor, ya que permiten obtener resultados en cuestión de horas, siendo este un factor crítico para poder establecer lo más pronto posible una pauta farmacológica adecuada. Sin embargo, el principal inconveniente es que, con excepción de la rifampicina en la que el 97% de las resistencias están codificadas por el gen rpoB, desconocemos las alteraciones genéticas que causan entre un 20 y un 40% de las resistencias al resto de fármacos de primera línea10; sin olvidar que su coste elevado, puede suponer un obstáculo para países con recursos económicos escasos.
El grado de incertidumbre de los métodos moleculares actuales justifica la necesidad de seguir realizando pruebas de sensibilidad fenotípicas, siendo además recomendable que la metodología empleada se ajuste a estándares de calidad internacionalmente aceptados, y que pueda ser verificada mediante controles de calidad externos que avalen la fiabilidad de los resultados obtenidos. En Asturias la participación en estos programas de control se inició en el año 2004.
Nuestro estudio pretende, como objetivo principal, valorar la aportación de las nuevas técnicas moleculares al diagnóstico de resistencias de M. tuberculosis comparándolas con el método fenotípico de referencia y conocer la situación de las resistencias a fármacos antituberculosos de primera línea en Asturias.
Material y métodoEstudio prospectivo que incluye la totalidad de las cepas de M. tuberculosis procedentes de muestras clínicas aisladas en Asturias durante el período comprendido entre enero de 2004 y diciembre de 2013.
Los criterios de inclusión de los aislamientos fueron: en pacientes con varias muestras del mismo origen, se incluyó el primer aislamiento de M. tuberculosis obtenido de cada paciente. En el caso de pacientes con muestras de diferentes orígenes, se incluyó una cepa de M. tuberculosis por cada localización de la tuberculosis.
Para las pruebas de sensibilidad se siguieron las directrices del Clinical and Laboratory Standards Institute11, realizándose el método de las proporciones múltiples de Canetti en Middlebrook 7H10 como método de referencia. Se analizaron las siguientes concentraciones críticas: isoniacida 0,2μg/ml, rifampicina 1μg/ml, etambutol 5μg/ml y estreptomicina 2μg/ml. Este método fue realizado tanto a las 1.861 cepas de M. tuberculosis incluidas en el estudio, como a los controles de calidad de la OMS recibidos de forma periódica durante la década estudiada.
En cuanto a la detección molecular de resistencias, en el caso de la isoniacida y rifampicina se realizó a todas las cepas incluidas en el estudio, mientras que para el etambutol y la estreptomicina se realizó solamente a las 102 cepas que presentaron resistencia fenotípica a algún fármaco de primera línea. Los métodos comerciales utilizados para las diferentes dianas fueron: rpoB, katG, inhA, gyrA, gyrB, rrs, eis (INNOLiPA RIFTB©; GenotypeMDRplus©; GenotypeMDRsl©).
Se recogió toda la información en una base de datos diseñada para tal fin con el programa Microsoft Excel. El análisis estadístico fue el recomendado para comparar una técnica diagnóstica con la que se obtienen resultados binarios o dicotómicos (prueba molecular: presencia/ausencia de mutación) con el gold standard (antibiograma fenotípico), construyendo tablas de contingencia para calcular: sensibilidad, especificidad, VPP, VPN.
ResultadosCasos de tuberculosis con confirmación microbiológica. Datos demográficosEntre enero de 2004 y diciembre de 2013 se diagnosticaron en Asturias un total de 1.826 casos de TB con confirmación microbiológica.
La incidencia fue mayor en hombres (68,3%) que en mujeres (31,7%), resultando una relación de masculinidad de 2,15 hombres por cada mujer.
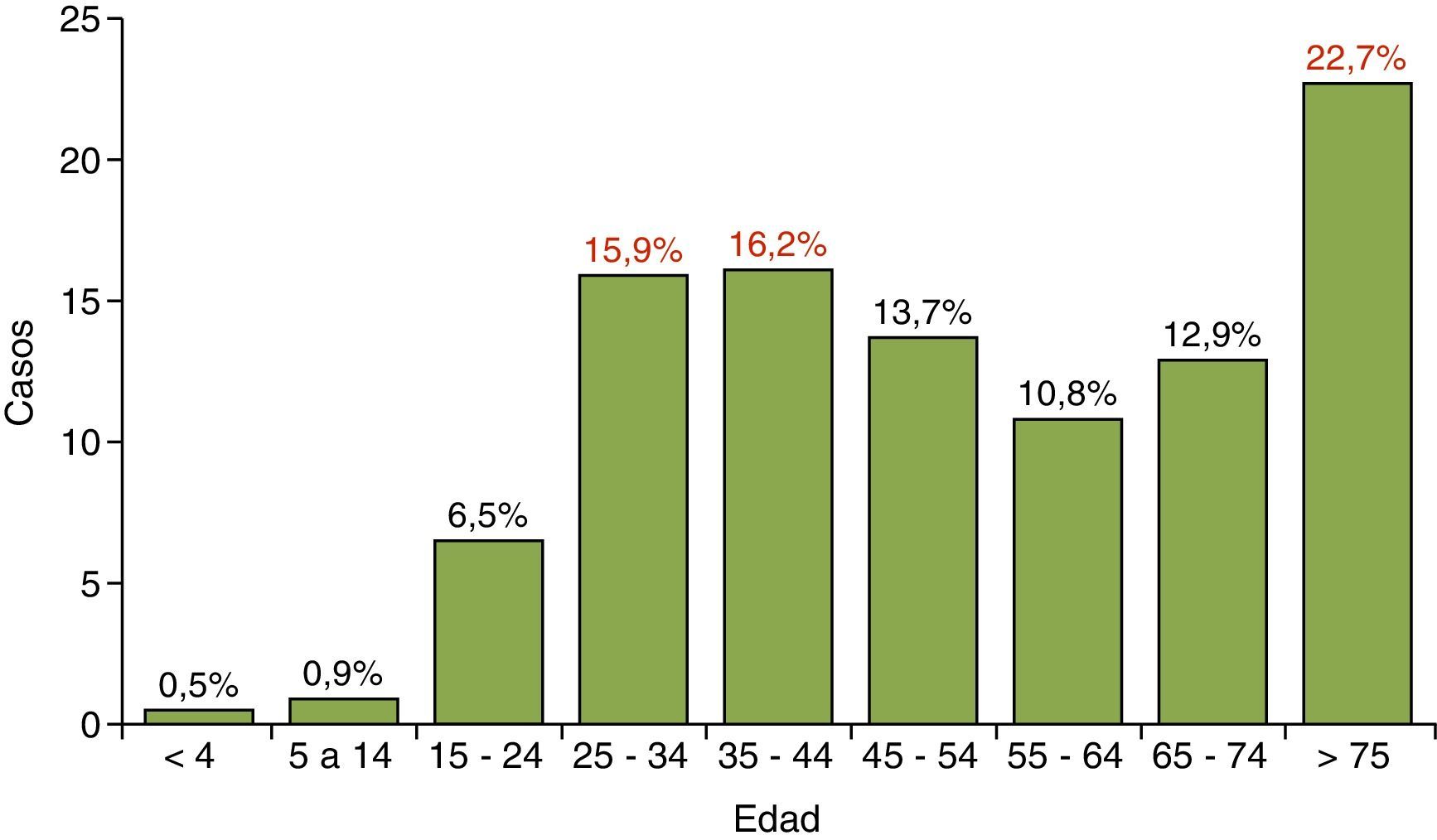
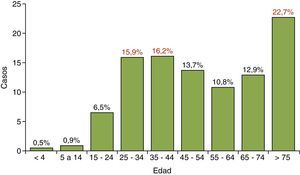
La media de edad fue de 53 años y los rangos de edad donde se concentraron la mayoría de los casos fueron los mayores de 75 años y las edades comprendidas entre 25 y 44 años, conformando la típica curva de distribución bimodal. El rango de edad que presentó menos casos de TB fue el de los menores de 15 años con 27 casos que representan el 1,4% del total, como se observa en la figura 1.
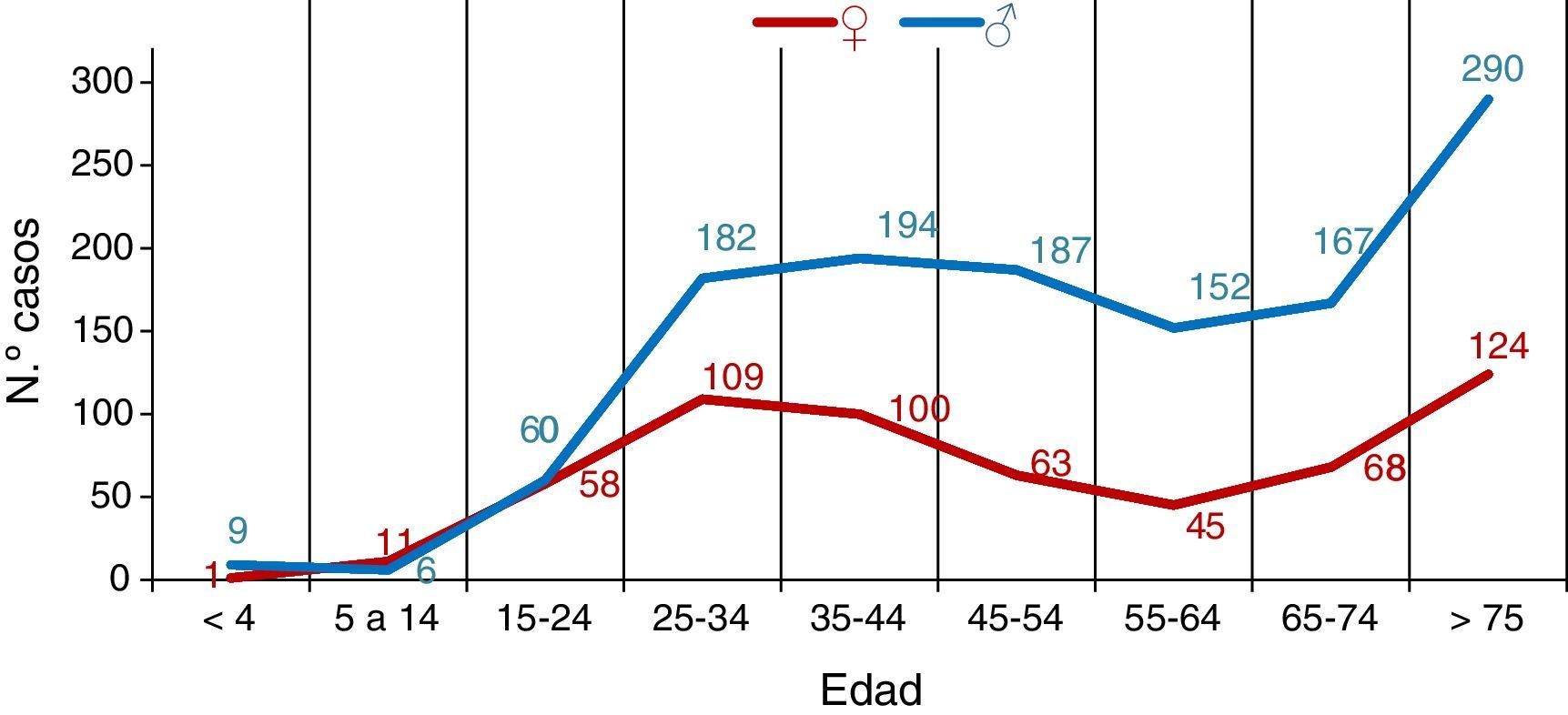
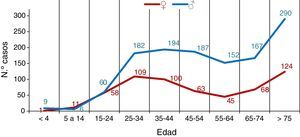
El número de casos de TB fue claramente superior en hombres en prácticamente todos los rangos de edad, excepto de 5 a 14 años en que el número de casos de TB fue superior en mujeres y de 15 a 24 años donde la incidencia se igualó en ambos sexos, como puede observarse en la figura 2.
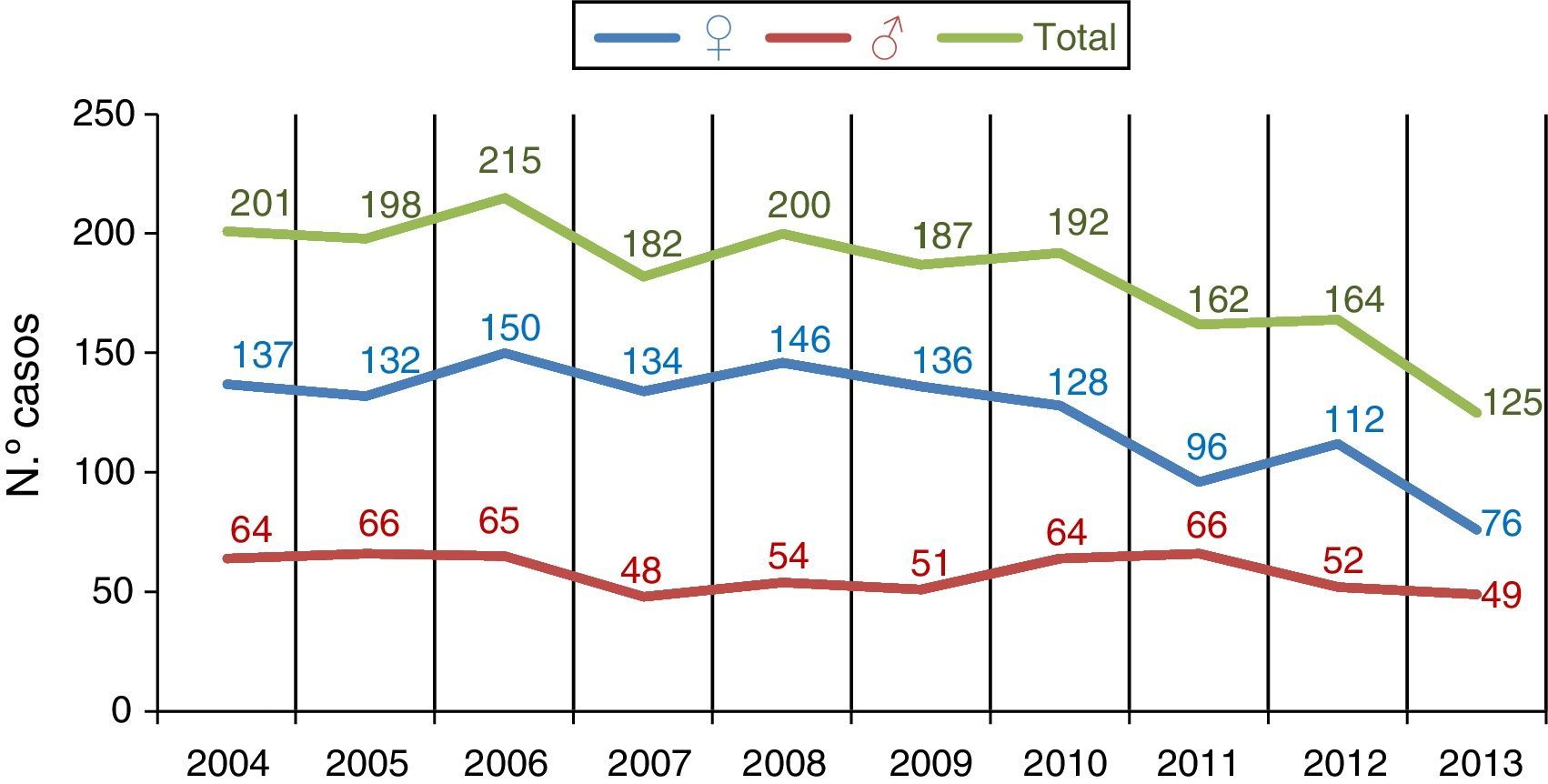
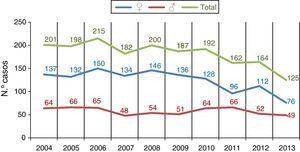
En la figura 3 se muestra el número de casos anuales de tuberculosis en hombres y en mujeres, observando que se produce un descenso moderado de casos que afecta principalmente a los hombres y aunque se produjo en todos los rangos de edad, fue más acentuado en edades comprendidas entre los 25 y 44 años.
Cepas de M. tuberculosis aisladas en Asturias (2004-2013)Un paciente puede tener TB en más de una localización anatómica, por este motivo los 1.826 casos anteriormente descritos se corresponden con 1.889 cepas de M. tuberculosis complex aisladas durante el período de estudio en la Unidad de Referencia Regional de Micobacterias de Asturias. (Se han excluido las cepas de Mycobacterium bovis (M. bovis) BCG aisladas en orina, en el contexto de pacientes con carcinoma transicional de vejiga tratados con instilaciones de BCG).
En cuanto a la distribución de las especies identificadas: 1.861 cepas (98,51%) correspondieron a M. tuberculosis hominis, 20 cepas (1,05%) fueron M. bovis bovis, 5 cepas (0,26%) Mycobacterium africanum tipo I y 3 cepas (0,15%) M. bovis BCG. No se identificó ninguna cepa del resto de especies que componen el complejo tuberculoso: Mycobacterium canetti, Mycobacterium caprae, Mycobacterium pinnipedi y Mycobacterium microti.
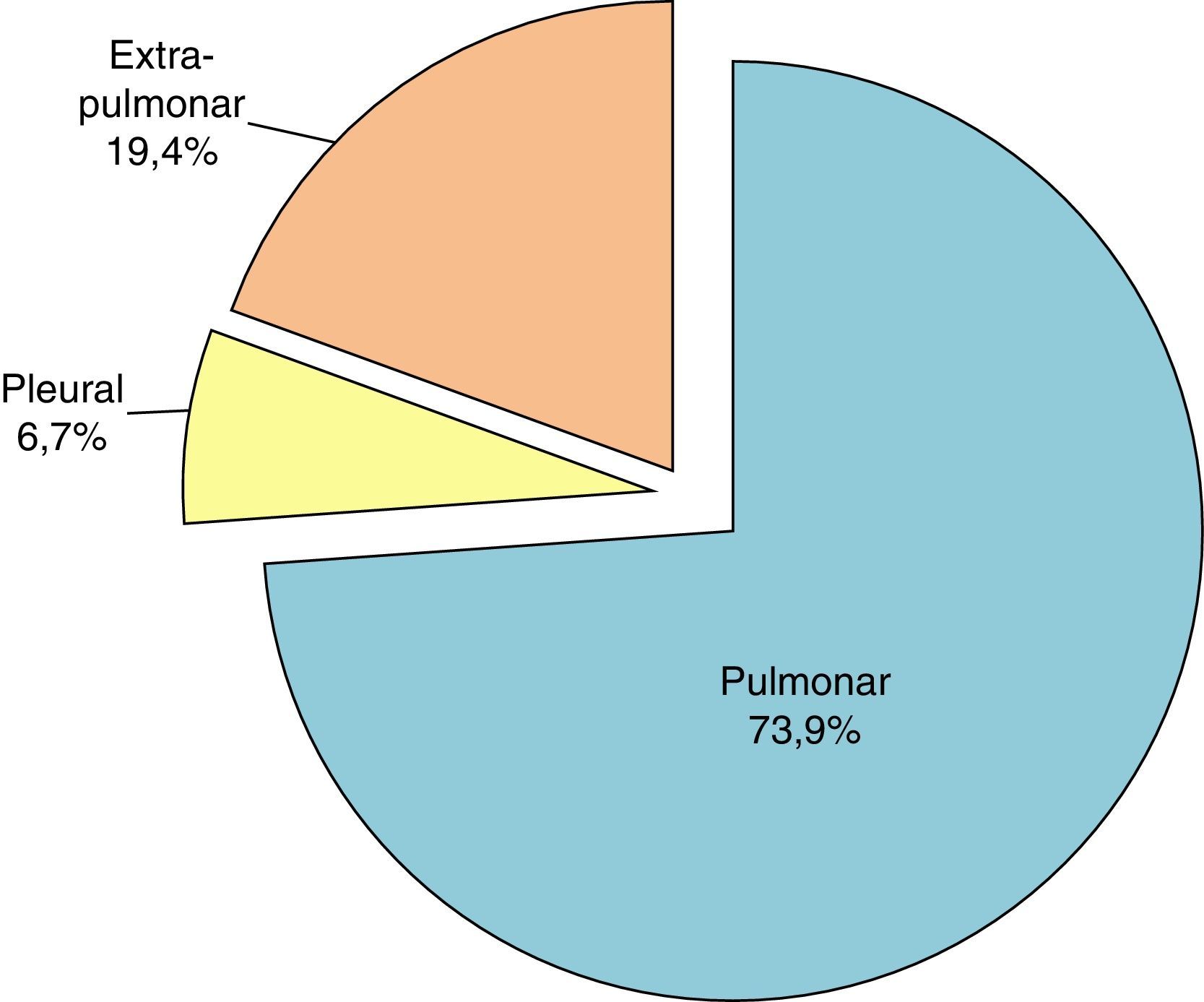
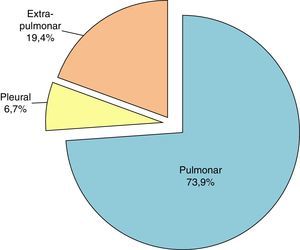
Origen de los aislamientos de M. tuberculosis hominisLa localización mayoritaria fue, como era de esperar, la pulmonar que supuso un 73,9% del total, como se puede apreciar en la figura 4.
Dentro de las tuberculosis extrapulmonares, la ganglionar fue la más frecuente con 65 casos (3,5%). Un análisis más detallado de la procedencia de las muestras incluidas en el estudio, se describe en la tabla 1. Las muestras procedentes de exudados purulentos, abscesos y úlceras de origen no especificado se clasificaron como «otras muestras extrapulmonares» y representaron un 10,8% del total.
Clasificación según la procedencia de las muestras incluidas en el estudio
| Procedencia de las muestras | N.° muestras | % del total |
|---|---|---|
| Pulmonar | 1.375 | 73,9 |
| Esputo | 1.196 | |
| Aspirado bronquial | 108 | |
| Lavado broncoalveolar | 34 | |
| Biopsia/PAAF pulmonar | 23 | |
| Lavado gástrico | 14 | |
| Pleural | 124 | 6,7 |
| Líquido pleural | 101 | |
| Biopsia/PAAF pleural | 23 | |
| Extrapulmonar | 362 | 19,4 |
| Ganglionar | 65 | 3,5 |
| Biopsia/PAAF ganglio | 65 | |
| Genitourinaria | 47 | 2,5 |
| Orina | 45 | |
| Biopsia epidídimo | 1 | |
| Biopsia endometrio | 1 | |
| Digestiva | 19 | 1,0 |
| Líquido ascítico | 16 | |
| Biopsia epiplón | 1 | |
| Biopsia colon | 1 | |
| Nódulo peritoneal | 1 | |
| Osteoarticular | 11 | 0,6 |
| Líquido sinovial | 4 | |
| Biopsia/punción ósea | 3 | |
| PAAF disco | 2 | |
| Biopsia sinovial | 1 | |
| Biopsia disco | 1 | |
| Pericárdica | 9 | 0,5 |
| Líquido pericárdico | 8 | |
| Biopsia pericardio | 1 | |
| Meníngea | 7 | 0,4 |
| LCR | 7 | |
| Otras extrapulmonares | 204 | 10,9 |
| Total | 1861 | 100 |
LCR: líquido cefalorraquídeo; PAAF: punción aspiración con aguja fina.
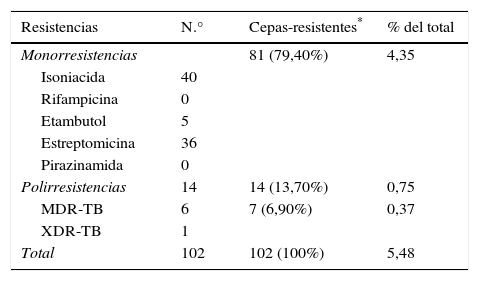
De las 1.861 cepas de M. tuberculosis hominis incluidas en el estudio, 102 cepas (5,48%) presentaban resistencias (iniciales y adquiridas) a algún fármaco de primera línea distribuidas del siguiente modo: 81 (4,35%) a un solo fármaco, 14 (0,75%) con polirresistencia (resistencia a más de un fármaco excluyendo la resistencia simultánea a isoniacida y rifampicina) y 7 (0,37%) MDR-TB (una de estas cepas era XDR-TB). Las 1.759 cepas restantes (94,52%) eran sensibles a todos los fármacos de primera línea.
En la tabla 2, se aprecia la distribución de los 102 aislamientos resistentes de M. tuberculosis hominis, en función de las resistencias a cada uno de los fármacos de primera línea.
Distribución de las resistencias de M. tuberculosis a fármacos de primera línea en Asturias
| Resistencias | N.° | Cepas-resistentes* | % del total |
|---|---|---|---|
| Monorresistencias | 81 (79,40%) | 4,35 | |
| Isoniacida | 40 | ||
| Rifampicina | 0 | ||
| Etambutol | 5 | ||
| Estreptomicina | 36 | ||
| Pirazinamida | 0 | ||
| Polirresistencias | 14 | 14 (13,70%) | 0,75 |
| MDR-TB | 6 | 7 (6,90%) | 0,37 |
| XDR-TB | 1 | ||
| Total | 102 | 102 (100%) | 5,48 |
MDR-TB: tuberculosis multirresistente; XDR-TB: tuberculosis extremadamente resistente.
De las 14 cepas con polirresistencia: 13 presentaban resistencia al menos a isoniacida, 4 a etambutol, 13 a estreptomicina, 7 a pirazinamida y ninguna a rifampicina. Por ese motivo el número total de resistencias a fármacos que suman 137 es mayor que el número de cepas resistentes que son 102.
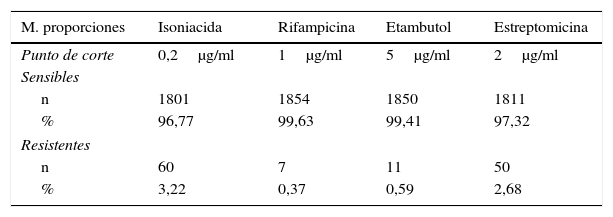
Pruebas de sensibilidad fenotípicas a fármacos de primera líneaUtilizando el método de las proporciones múltiples como referencia, las cepas se clasificaron como sensibles o resistentes para cada fármaco, según los diferentes puntos de corte previamente descritos, tal y como se muestra en la tabla 3.
Clasificación de las cepas como sensibles o resistentes según el método de referencia
| M. proporciones | Isoniacida | Rifampicina | Etambutol | Estreptomicina |
|---|---|---|---|---|
| Punto de corte | 0,2μg/ml | 1μg/ml | 5μg/ml | 2μg/ml |
| Sensibles | ||||
| n | 1801 | 1854 | 1850 | 1811 |
| % | 96,77 | 99,63 | 99,41 | 97,32 |
| Resistentes | ||||
| n | 60 | 7 | 11 | 50 |
| % | 3,22 | 0,37 | 0,59 | 2,68 |
También se detectaron 9 cepas resistentes a pirazinamida, pero este antibiótico no fue incluido en el estudio por no ser las proporciones múltiples un método de referencia para la realización de pruebas de sensibilidad a este fármaco.
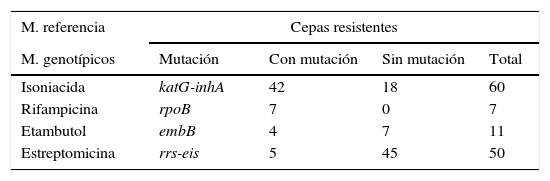
Detección molecular de resistencias a fármacos de primera líneaLa frecuencia de las mutaciones detectadas a cada fármaco de primera línea en cepas resistentes y en cepas sensibles por el método de las proporciones múltiples, se desglosa en la tabla 4, pudiendo observar cómo en 20 cepas la detección de mutaciones no conllevaba resistencia fenotípica, aunque conviene reseñar que en 16 de ellas se correspondía con una mutación silente en el codón 514 del gen rpoB que no implica resistencia a la rifampicina.
Detección molecular de resistencias a fármacos de primera línea, en cepas resistentes y en cepas sensibles por el método fenotípico de referencia
| M. referencia | Cepas resistentes | |||
|---|---|---|---|---|
| M. genotípicos | Mutación | Con mutación | Sin mutación | Total |
| Isoniacida | katG-inhA | 42 | 18 | 60 |
| Rifampicina | rpoB | 7 | 0 | 7 |
| Etambutol | embB | 4 | 7 | 11 |
| Estreptomicina | rrs-eis | 5 | 45 | 50 |
| M. referencia | Cepas sensibles | |||
|---|---|---|---|---|
| M. genotípicos | Mutación | Con mutación | Sin mutación | Total |
| Isoniacida | katG-inhA | 3 (inhA) | 1798 | 1801 |
| Rifampicina | rpoB | 16 (rpoB514) | 1838 | 1854 |
| Etambutol | embB | 1 | 90 | 91 |
| Estreptomicina | rrs-eis | 0 | 52 | 52 |
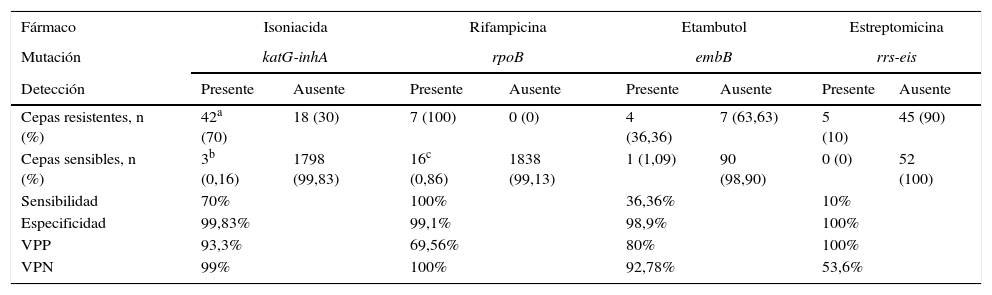
La detección molecular de resistencias a cada uno de los fármacos y su concordancia/discordancia con los métodos fenotípicos, se puede apreciar en la tabla 5. La capacidad de los métodos genotípicos para detectar mutaciones en nuestra serie iba desde un 100% en el caso de la rifampicina, hasta el 10% en la estreptomicina. Los porcentajes de especificidad de los métodos genotípicos fueron muy altos, entre un 98,90 y el 100%.
Detección molecular de resistencias a fármacos, concordancia/discordancia con respecto a las pruebas de sensibilidad fenotípicas
| Fármaco | Isoniacida | Rifampicina | Etambutol | Estreptomicina | ||||
|---|---|---|---|---|---|---|---|---|
| Mutación | katG-inhA | rpoB | embB | rrs-eis | ||||
| Detección | Presente | Ausente | Presente | Ausente | Presente | Ausente | Presente | Ausente |
| Cepas resistentes, n (%) | 42a (70) | 18 (30) | 7 (100) | 0 (0) | 4 (36,36) | 7 (63,63) | 5 (10) | 45 (90) |
| Cepas sensibles, n (%) | 3b (0,16) | 1798 (99,83) | 16c (0,86) | 1838 (99,13) | 1 (1,09) | 90 (98,90) | 0 (0) | 52 (100) |
| Sensibilidad | 70% | 100% | 36,36% | 10% | ||||
| Especificidad | 99,83% | 99,1% | 98,9% | 100% | ||||
| VPP | 93,3% | 69,56% | 80% | 100% | ||||
| VPN | 99% | 100% | 92,78% | 53,6% | ||||
VPN: valor predictivo negativo; VPP: valor predictivo positivo.
La capacidad de detección de cepas resistentes de los métodos moleculares, varía mucho en función del conocimiento de los genes implicados en la resistencia a cada uno de los fármacos y la inclusión de esas dianas en los métodos comerciales, siendo en el caso de la rifampicina una herramienta excelente para la detección y sospecha inicial de cepas multirresistentes. El análisis de las cepas de M. tuberculosis aisladas en Asturias durante la década 2004 a 2013, nos revela que la tasa de resistencias a fármacos de primera línea es baja y los casos de multirresistencia son esporádicos.
En nuestra serie, al igual que en otros estudios publicados6, se observa un predominio en hombres, resultando una proporción de 2,1 hombres por cada mujer, cifra superior a la media española de 1,7.
La media de edad fue de 53 años, superior a la media española que es de 44 años5. El grupo de edad que presentó menos casos de TB fue el de los menores de 15 años con 27 casos lo que representa el 1,4% del total12.
Como era de esperar, M. tuberculosis hominis fue la especie más frecuentemente identificada con 1.861 cepas que representan el 98,5%. La localización pulmonar fue la más frecuente 73,9% y la pleural un 6,7%, porcentajes algo superiores a los nacionales donde el 70% fueron de localización pulmonar y un 4,9% pleural13.
El control adecuado de la TB precisa conocer periódicamente la prevalencia de las resistencias de M. tuberculosis a fármacos antituberculosos, sin embargo la magnitud de la resistencia a estos fármacos en España es difícil de calcular ya que se dispone de pocos estudios publicados, y los que hay ofrecen cifras muy variadas de resistencias14. En 2014 se publica el primer estudio prospectivo de resistencias a fármacos antituberculosos de primera línea en una amplia cohorte de pacientes15. Nuestros datos de resistencias globales (iniciales y adquiridas) a uno o más fármacos de primera línea en cepas de M. tuberculosis han sido del 5,5%, inferiores a las publicadas en este estudio (9,2%).
Las tasas de resistencia a cada uno de los fármacos de primera línea fueron inferiores a la media española: isoniacida (3,22 vs. 6,7%); rifampicina (0,37 vs. 1,9%), etambutol (0,59 vs. 1%), estreptomicina (2,68 vs. 4,4%), no se detectó ningún caso con monorresistencia a rifampicina. Así mismo el porcentaje de cepas MDR-TB también fue inferior a la media nacional (0,37 vs. 1,2%). Teniendo en cuenta la relación directa encontrada entre la población inmigrante y los casos de MDR-TB16, la escasa población inmigrante en Asturias (10,4%)17 con respecto a otras comunidades españolas, podría ser un factor determinante.
En lo referente a los métodos moleculares disponibles comercialmente, conviene recordar que no incluyen todas las dianas relacionadas con la resistencia, o bien no contemplan la variabilidad existente en cuanto a los tipos de mutaciones prevalentes en cada área geográfica10. En nuestra serie, se observa una especificidad muy alta que puede ser debida a que se basan en el análisis simultáneo de la mutación y de la secuencia silvestre correspondiente18, sin embargo la sensibilidad es muy variable dependiendo del fármaco analizado.
Se ha demostrado que la resistencia a rifampicina en M. tuberculosis se explica en un 96 a 98% por mutaciones en el gen rpoB19, este hecho facilita enormemente el desarrollo de técnicas moleculares para la detección rápida de la resistencia a la rifampicina en la cepa aislada por cultivo o, incluso, directamente en la muestra clínica.
En el caso de la rifampicina, todos los casos de resistencia fenotípica fueron detectados mediante las técnicas moleculares, correspondiendo los 7 casos a cepas multirresistentes. Por lo tanto estas técnicas ofrecen un gran potencial en la predicción de multirresistencia y permiten establecer desde un inicio una orientación del tratamiento farmacológico más adecuado.
Sin embargo, ni siquiera en el caso de la rifampicina los métodos moleculares deberían excluir a los fenotípicos ya que, entre las cepas analizadas 16 de ellas presentaban una mutación silente TTC/TTT en el codón 514 del gen rpoB que no implicaba resistencia fenotípica20. Estos resultados han sido hallados también por otros grupos de estudio, usando otros métodos moleculares basados en la detección de mutaciones en el gen rpoB21–23.
En el caso de la resistencia a la isoniacida en M. tuberculosis está asociada a múltiples genes (katG, inhA, aphC, ndh, kasA, furA, ethA), siendo los dos primeros los implicados más frecuentemente (60-65% de los casos)18, con importantes variaciones geográficas. De las 1.861 cepas incluidas en nuestra serie, 60 cepas fueron resistentes a la isoniacida por el método de referencia y de ellas, 42 cepas (70%) presentaron mutaciones en los genes katG y/o inhA. El 30% de las cepas resistentes a la isoniacida no presentaba alteraciones del gen katG ni del inhA. Existe la posibilidad de que otros genes involucrados en la resistencia a isoniacida, pero no incluidos en los sistemas comerciales, expliquen el mecanismo de resistencia en estas cepas10.
En cuanto a los restantes fármacos de primera línea, entre el 50-70% de las cepas resistentes a etambutol, contienen una mutación en la región determinante de la resistencia a etambutol (ethambutol resistance determining región) del gen embB24. En la actualidad, los sistemas comerciales para la detección rápida de mutaciones relacionadas con la resistencia al etambutol se basan solo en la diana embB306.
En nuestro estudio, de las 11 cepas que fueron resistentes al etambutol por el método de referencia, 4 cepas (36,36%) presentaron mutaciones en el gen embB y 7 cepas (63,64%) no mostraron ninguna de estas mutaciones.
En el caso de la estreptomicina, se hallan mutaciones en los genes rrs y rpsL en el 55% de los aislamientos resistentes a este fármaco18. La mayoría de las mutaciones puntuales ocurren en el gen rpsL, siendo la mutación más frecuente la que ocurre en el codón 43, que se relaciona con una resistencia de alto nivel; mientras que las que afectan al gen rrs producen una resistencia de nivel intermedio-bajo25 y se hallan principalmente en los codones 530 y 91226–29. Sin embargo, existen cepas con bajos niveles de resistencia fenotípica que no poseen mutación alguna en estos genes, lo que hace postular el papel de otras dianas alternativas, como el gen gidB (que codifica una metiltransferasa específica para el 16S ARNr)30 o incluso bombas de expulsión31.
En el caso de la estreptomicina existen limitaciones en la detección mediante métodos comerciales de mutaciones que producen resistencia, ya que no están incluidas las que se producen en el gen rpsL. En nuestra serie de las 50 cepas que fueron resistentes por el método de referencia, 5 cepas (10%) presentaron mutaciones en el gen rrs-eis y 45 cepas (90%) no mostraron ninguna de estas mutaciones.
Las técnicas moleculares aportan rapidez en la obtención de resultados, sin embargo su sensibilidad varía mucho en función del fármaco estudiado, siendo excelente en el caso de la rifampicina. En ningún caso estas técnicas deberían sustituir a las pruebas de sensibilidad fenotípicas convencionales, ya que una mutación genética no implica en todos los casos una resistencia fenotípica20, así como tampoco la ausencia de mutaciones en los genes citados excluye totalmente la posibilidad de resistencias fenotípicas. De cara al futuro, resulta necesario intensificar la investigación para identificar nuevos mecanismos de resistencia en M. tuberculosis, así como perfeccionar la metodología actualmente existente con la incorporación progresiva de las nuevas dianas identificadas, como en el caso del gen rpsL y la estreptomicina; de los nuevos fármacos para el tratamiento de la tuberculosis (fluoroquinolonas, linezolid, bedaquilina, delamanid, etc.); e idealmente que la detección de mutaciones en todos los genes diana se pudiera hacer de manera simultánea.
ConclusionesLas técnicas moleculares aportan rapidez en la obtención de resultados, sin embargo su sensibilidad varía mucho en función del fármaco estudiado, por lo que los resultados deben confirmarse con las pruebas de sensibilidad fenotípicas de referencia. La tasa de resistencias a fármacos de primera línea en Asturias es baja e inferior a otras zonas de nuestro país y los casos de multirresistencia son esporádicos.
Autoría/colaboradores- -
Juan José Palacios Gutiérrez: director del trabajo y responsable de la Unidad de Referencia Regional de Micobacterias del Servicio de Microbiología del HUCA.
- -
Luz María Alba Álvarez: análisis de datos y redacción.
- -
José María García García: Unidad de Gestión Clínica de Neumología, Hospital San Agustín, Avilés: corrección y supervisión del trabajo.
- -
María Dolores Pérez Hernández: Servicio de Vigilancia y Alertas Epidemiológicas, Dirección General de Salud Pública, Consejería de Sanidad: aportación y revisión de datos demográficos.
- -
Susana Martínez González: revisión bibliográfica.
- -
Red de laboratorios de Microbiología del SESPA: aislamiento de las cepas remitidas a la Unidad de Referencia de Micobacterias del HUCA.
Los autores declaran no tener ningún conflicto de intereses.
Ángela Menéndez González, Macarena Álvarez Fernández, Ángeles Díaz Escalada por la perfección con la que llevan a cabo su trabajo diario.
La Red de Laboratorios de Microbiología del SESPA incluye los Servicios de Microbiología de: Hospital de Jarrio, Hospital Carmen y Severo Ochoa, Hospital San Agustín, Hospital de Cabueñes, Hospital de Jove, Hospital Grande Covián, Hospital Álvarez-Buylla y Hospital Valle del Nalón.