El déficit hereditario de alfa 1-antitripsina (α1-AT) predispone al desarrollo de enfisema pulmonar. El objetivo de este trabajo es evidenciar las limitaciones de algunos métodos de laboratorio utilizados en el estudio del déficit, cuya interpretación puede inducir a error en la detección de alelos poco frecuentes.
Se describen dos casos clínicos: la paciente índice, que presentaba enfisema pulmonar con niveles de α1-AT inferiores a 12mg/dL, catalogada erróneamente como homocigota de la variante alélica normal PI MM mediante un método de genotipificado rápido; la madre de la paciente, asintomática, con niveles bajos (60mg/dL), catalogada también como PI MM.
La secuenciación del gen catalogó a la paciente índice como portadora del alelo nulo PI Clayton y del deficitario PI Mmalton. La madre resultó portadora heterocigota PI Clayton/PI M.
Los resultados ponen de relieve las dificultades de diagnóstico del déficit así como la necesidad de consensuar métodos para este estudio.
Hereditary alpha-1-antitrypsin (α1-AT) deficiency predisposes to pulmonary emphysema. The objective of this study is to demonstrate the limitations of some laboratory methods used in the study of the deficiency, and which may produce errors in interpretation and detection of uncommon alleles.
Two clinical cases are described: the index patient, who had pulmonary emphysema with α1-AT levels less than 12mg/dL, was erroneously classified as a homozygote of the normal allelic variant PI MM using a rapid genotype method; the mother of the patient, asymptomatic, with low levels (60mg/dL), was also classified as PI MM.
The gene sequencing classified the index patient as a carrier of the PI Clayton null allele and PI Mmalton deficient. The mother was a PI Clayton/PI heterozygote carrier.
These results highlight the difficulties in diagnosing the deficiency, as the well as the need to reach a consensus on methods for this study.
El déficit de alfa-1-antitripsina (α1-AT) es un trastorno hereditario asociado con enfisema pulmonar en adultos1. La principal función fisiológica de α1-AT, también conocida como inhibidor de las proteinasas (PI), es proteger el tejido pulmonar de las enzimas proteolíticas2. Los alelos más comunes asociados con niveles normales de α1-AT son en general los PI M. Sin embargo, además de los alelos PI normales existen también alelos PI M, poco frecuentes, que son deficitarios3,4. Los alelos más comunes asociados con niveles bajos de α1-AT son el PI S y el PI Z, siendo la mayoría de los individuos con deficiencia grave los homocigotos PI Z5,6. La deficiencia también puede deberse a otros alelos, disfuncionales o nulos7,8.
El diagnóstico de deficiencia implica la cuantificación de α1-AT y el posterior fenotipificado o genotipificado cuando la concentración es baja9. Sin embargo, algunos métodos de genotipificado rápido conducen fácilmente a diagnósticos erróneos, ya que algunos alelos poco frecuentes y en particular los nulos, no son detectados. Es el caso de las denominadas técnicas alelo-específico que únicamente detectan los alelos PI S y PI Z10–13.
En esta observación clínica se reflejan los errores en el diagnóstico en dos casos con déficit de α1-AT portadores heterocigotos del alelo nulo PI Clayton, que habían sido considerados homocigotos normales PI MM mediante un método de hibridación alelo-específico inadecuadamente interpretado.
Observación clínicaSe incluyeron dos miembros de la misma familia: El caso índice es una mujer de 35 años, exfumadora. Presentaba una clínica de hiperreactividad bronquial asociada a procesos infecciosos de vías aéreas inferiores. La determinación de los niveles de α1-AT fue claramente deficitaria: 12mg/dL (rango de normalidad: 110-190mg/dL).
En la TAC torácica se observaban signos de enfisema difuso panacinar con mayor afectación en lóbulos inferiores y signos de enfisema paraseptal en ambos ápex y lóbulos superiores. Las pruebas funcionales con tratamiento broncodilatador fueron: FVC 3,39 L (93% pred), FEV1 1,34 L (42% pred), FEV1/FVC 40, TLC 7,34 (138% pred), RV 3,95 L (247% pred), DLCO 5,9 (64% pred), DLCO/VA 1,11 (53% pred).
Debido a la baja concentración de α-1AT no se pudo determinar el fenotipo. Por esta razón se optó por determinar el genotipo mediante un método de hibridación alelo-específico que sólo detecta las variantes PI S y PI Z. En este análisis el paciente fue catalogado como no portador de estos alelos, y por exclusión se reportó erróneamente como portador de la variante normal más común de α1-AT, la PI MM. La rápida progresión clínica y el bajo nivel de α1-AT indujo a iniciar tratamiento sustitutivo (180mg/kg/21 días de α1-AT) mientras se investigaba por secuenciación la discordancia entre los niveles y el genotipo asignado.
La madre de la paciente, de 71 años, asintomática desde el punto de vista respiratorio, presentaba una α1-AT de 60mg/dL y había sido catalogada también, por el mismo método de hibridación alelo-específico, como portadora homocigota PI MM. Esta incoherencia indujo también el estudio del genotipo.
En ambos casos la determinación del genotipo de α1-AT se llevó a cabo en ADN aislado de sangre total mediante un procedimiento de amplificación y secuenciación14.
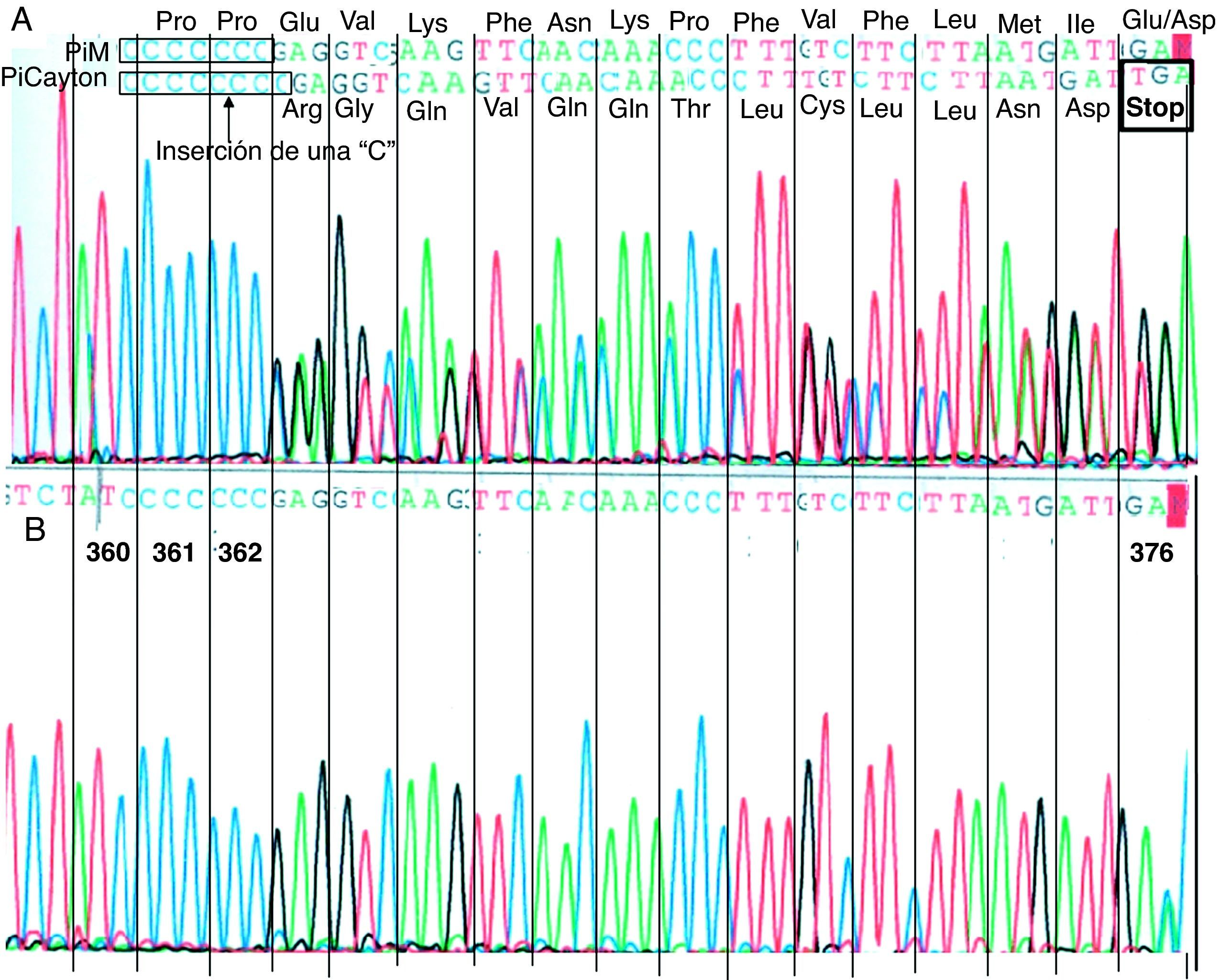
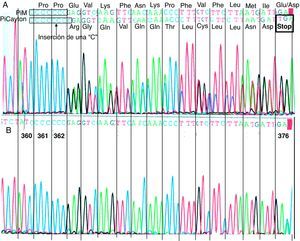
El análisis de la secuencia de ADN mostró que el caso índice presentaba dos diferentes mutaciones correspondientes a las variantes alélicas PI Clayton y PI Mmalton: la PI Clayton es una variante nula de α1-AT caracterizada por la inserción de una citosina en el exón V del gen15. Como consecuencia de esta inserción a la proteína le faltan 19 aminoácidos. En la figura 1 se muestra un fragmento del exón V en la que se observa la mutación correspondiente a la variante PI Clayton. La PI Mmalton es una variante deficitaria, poco frecuente, caracterizada por la deleción de 3 pares de bases (TTC) en el exón II16. La variante PI Mmalton está asociada a una concentración de α1-AT similar a la observada en un individuo PI ZZ (de 15-35mg/dL, aproximadamente). A la paciente se la consideró como heterocigota de las dos variantes deficitarias.
Secuenciación directa de un fragmento del exón V del gen de la α1-AT en la región correspondiente al alelo PI Clayton. La PI Clayton es una variante nula de α1-AT caracterizada por la inserción de una citosina en el exón V de la secuencia alélica M1 normal (Val213) que ya normalmente tiene siete citosinas (C), que codifican los aminoácidos 360 a 362. A) secuencia correspondiente al caso índice, heterocigoto para la variante PI Clayton. En la parte superior de la figura se indican las dos secuencias solapadas: PI M (secuencia sin mutar) con siete citosinas y PI Clayton con ocho citosinas. B) secuencia de referencia del mismo fragmento, correspondiente a un paciente sin la mutación.
El análisis familiar mostró que la madre era heterocigota del alelo nulo PI Clayton y del alelo normal PI M.
DiscusiónLos alelos nulos de α1-AT se definen como genes en los que por una mutación en la secuencia de ADN no se expresa la proteína7,17,18. Son alelos extremadamente raros, de los que se han descrito aproximadamente unas 16 clases8. Nuestro equipo, que como laboratorio del Registro Español de Déficit de α1-AT lleva estudiando esta enfermedad desde el año 1993, únicamente había detectado un caso correspondiente a un paciente con EPOC portador del alelo nulo PI Bellingham. La importancia clínica de los alelos nulos se hace más evidente cuando se heredan ya sea con otro alelo nulo o con otro deficitario.
En esta observación clínica se describe el caso de una paciente con antecedentes de enfisema pulmonar grave, en tratamiento sustitutivo con α1-AT intravenosa, portadora heterocigota del alelo nulo PI Clayton. Por los datos disponibles, es el primer caso conocido de un paciente portador heterocigoto del alelo nulo PI Clayton juntamente con el alelo deficitario, poco frecuente, PI Mmalton, asociado con la degradación intracelular de α1-AT16. La madre de la paciente, asintomática, era heterocigota del alelo PI Clayton y del normal PI M. Los dos pacientes habían sido catalogados erróneamente como portadores homocigotos del alelo normal PI M. El nulo PI Clayton ha sido detectado anteriormente asociado con enfisema pulmonar, también en estado de heterocigosis con variantes deficitarias de α1-AT poco comunes, en EE.UU.18 y en Japón19.
En los casos clínicos analizados se ponen de relieve algunos de los problemas potenciales asociados con el diagnóstico del déficit de α1-AT. Debido a que el caso índice tenía unos niveles de α1-AT en suero muy bajos no se pudo determinar el fenotipo de α1-AT. En consecuencia, se optó por el análisis del genotipo mediante una técnica de hibridación alelo-específica que sólo detecta las variantes PI S y la PI Z. La paciente fue catalogada como no portadora de estos dos alelos y por exclusión como portadora de los alelos normales PI MM, al considerarse que si en la población general los alelos mayoritarios son PI M, PI S y PI Z, la combinación PI MM sería la más probable. Un error similar ocurrió en el genotipo de la madre. Las técnicas alelo-específicas son técnicas rápidas, fácilmente automatizables, de bajo coste pero de genotipificado parcial, es decir de detección de solo algunos alelos11. Únicamente informan de la presencia o ausencia de los alelos estudiados pero nunca se puede deducir, aunque sea lo más probable, que al no identificar estos alelos estemos ante un alelo normal. Estos métodos son fiables, siendo los errores relacionados principalmente con la incorrecta interpretación de los resultados.
Con el objetivo de facilitar el establecimiento de programas eficaces para detectar el mayor número de pacientes afectados de déficit de α1-AT se han publicado una serie de directrices9,20. La determinación de los niveles de α1-AT y del fenotipo en suero son los análisis de laboratorio recomendados. A cada fenotipo le corresponde un rango de valores de α1-AT determinado. En los casos en los que haya una discordancia entre los niveles y el fenotipo se debe realizar la caracterización molecular del gen mediante secuenciación. Hay que hacer hincapié en que la principal aplicación de las técnicas de detección alelo-específicas es el cribado de los alelos PI S y PI Z.
Por todo ello es importante que cada laboratorio informe del método utilizado en el estudio del déficit y de sus limitaciones si las tuviera, así como de los valores de referencia que corresponden a cada fenotipo o genotipo. Teniendo en cuenta que la mayoría de los hospitales disponen de equipos de secuenciación hay que abogar por esta técnica para el estudio molecular de todo el gen de α1-AT en los casos que el diagnóstico plantee dudas.