Los microARN (miRs) son pequeñas moléculas de ARN no codificante capaces de regular negativamente la expresión génica. Participan activamente en la modulación de importantes procesos celulares fisiológicos y están involucrados en la patogenia de enfermedades pulmonares tales como el cáncer de pulmón, la fibrosis pulmonar, el asma y la enfermedad pulmonar obstructiva crónica. Un mayor conocimiento del papel que los miRs desarrollan en estas patologías podría abrir las puertas a nuevas herramientas diagnósticas y terapéuticas. En esta revisión, repasamos el rol que algunos miRs desempeñan en la patogenia de ciertas enfermedades pulmonares, así como la posible proyección de estos descubrimientos hacia la aplicación clínica.
MicroRNAs (miRNAs) are small non-coding RNA molecules that negatively regulate gene expression. They actively participate in the modulation of important cell physiological processes and are involved in the pathogenesis of lung diseases such as lung cancer, pulmonary fibrosis, asthma and chronic obstructive pulmonary disease. A better understanding of the role that miRNAs play in these diseases could lead to the development of new diagnostic and therapeutic tools. In this review, we discuss the role of some miRNAs in different lung diseases as well as the possible future of these discoveries in clinical applications.
A lo largo de las últimas décadas, el análisis proteómico y genómico ha permitido comprender mejor la patogenia de ciertas enfermedades pulmonares. No obstante, a medida que se avanza en el conocimiento de su fisiopatología, la complejidad del sistema y el número de actores involucrados parecen incrementarse. Tras la exposición a una noxa diversos procesos celulares y mediadores intervienen antes de que la enfermedad se manifieste. En este sentido, los microARN (miRs) parecen jugar un rol importante en el desarrollo y progresión de las patologías pulmonares1–6.
Los miRs son una clase de ARN no codificante, evolutivamente conservados y de pequeño tamaño capaces de regular la expresión génica por distintos mecanismos7. Si bien pasaron varios años entre el descubrimiento del primer miR (lin-4) en 1993 y la caracterización del segundo miembro de la familia, hasta la fecha la cantidad de miRs descritos y el número de publicaciones científicas vinculadas a los mismos han crecido exponencialmente8. Cerca de 2000 miRs humanos han sido descritos (Sanger miRBase versión 18), estando involucrados en el control de importantes procesos fisiológicos y en la patogenia de diversas enfermedades5,7,9.
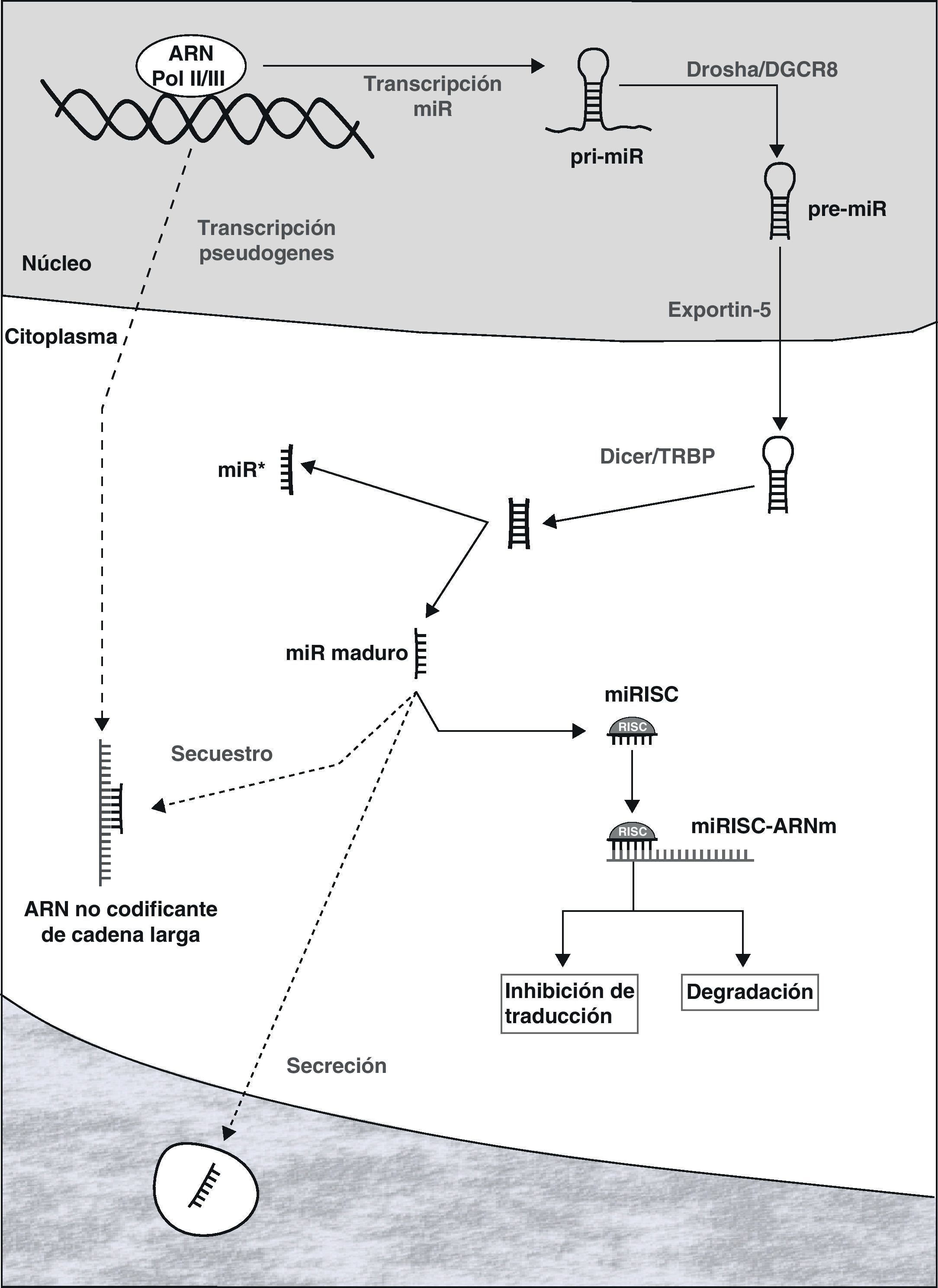
Biogénesis y función de los microARNLos miRs pueden localizarse en intrones o exones de genes codificantes de proteínas, o en sectores no codificantes del genoma. Una vez activada, la transcripción es llevada a cabo por la ARN polimerasa II o III, originando una estructura de ARN en forma de horquilla que constituye el transcripto primario del miR (pri-miR, figura 1)7. Dentro del núcleo, ambos extremos del pri-miR son cortados por el complejo Drosha/DGCR8 dando lugar al precursor del miR maduro (pre-miR), con un tamaño aproximado de 70-100 nucleótidos. Este es transportado activamente al citoplasma a través de Exportin-5, donde es procesado por el complejo Dicer/TRBP originando una molécula de ARN de doble cadena de 19 a 25 nucleótidos de largo. Una de las cadenas constituye el miR maduro, mientras que su complementario (denominado miR*) es generalmente degradado. El miR maduro es incorporado al complejo de silenciamiento inducido por ARN (RISC por sus siglas en inglés, RNA induced silencing complex), cuyos componentes más importantes son las proteínas de la familia Argonauta10.
Representación esquemática de las diferentes etapas en la biogénesis de los miRs y mecanismos de represión de la expresión génica: degradación del ARNm o inhibición de su traducción. Se muestra también la posible secreción de miRs, así como su interacción con moléculas de ARN no codificante de cadena larga.
Los miRs son fundamentalmente represores de la expresión génica a nivel postranscripcional, mediante la degradación del ARN mensajero (ARNm) o la inhibición de su traducción proteica11. Una vez que el miR se encuentra incorporado al RISC (miRISC), el complejo es capaz de reconocer al ARNm diana mediante la interacción entre regiones complementarias del miR y el ARNm. Si bien existen excepciones, como regla general el sitio de unión se encuentra en el extermo 3’ de la región no codificante del ARNm. El grado de complementariedad entre ambos parece ser uno de los determinantes del mecanismo de represión que se pone en marcha: degradación del ARNm si la misma es casi perfecta, o inhibición de la traducción cuando la complementariedad es menor. Si bien se creía que la degradación del ARNm era un mecanismo infrecuente en células animales, se ha puesto de manifiesto en la actualidad un rol importante del mismo12. Es relevante el hecho de que determinado ARNm puede ser diana de un gran número de miRs, de la misma forma que cada miR puede reprimir a decenas/centenares de genes. Además, existe evidencia que sugiere la posible transferencia de miRs de una célula a otra, generando un interesante mecanismo de comunicación y regulación intercelular13–15.
La actividad de los miRs se encuentra regulada de forma estricta, mediante el control de su transcripción, de las diferentes etapas de biogénesis y posteriormente de la función del miRISC16,17. Recientemente, se ha descrito la existencia de una compleja red de comunicación entre largas moléculas de ARN no codificante (producto de la transcripción de pseudogenes), miR y ARNm18,19. Estas moléculas de ARN no codificante (de cientos a miles de nucleótidos) poseen sitios de unión para miRs específicos, y se comportan como verdaderos «atrapadores» de miRs, impidiendo que los mismos repriman a sus ARNm diana.
Teniendo en cuenta la enorme capacidad de los miRs para influir sobre la expresión de gran parte del genoma y el intrincado sistema de control en el que se encuentran inmersos, es comprensible que la desregulación de los mismos repercuta significativamente sobre la homeostasis del organismo. Un número creciente de miRs ha sido implicado en distintas enfermedades pulmonares, permitiendo una mejor comprensión de la patogenia de las mismas.
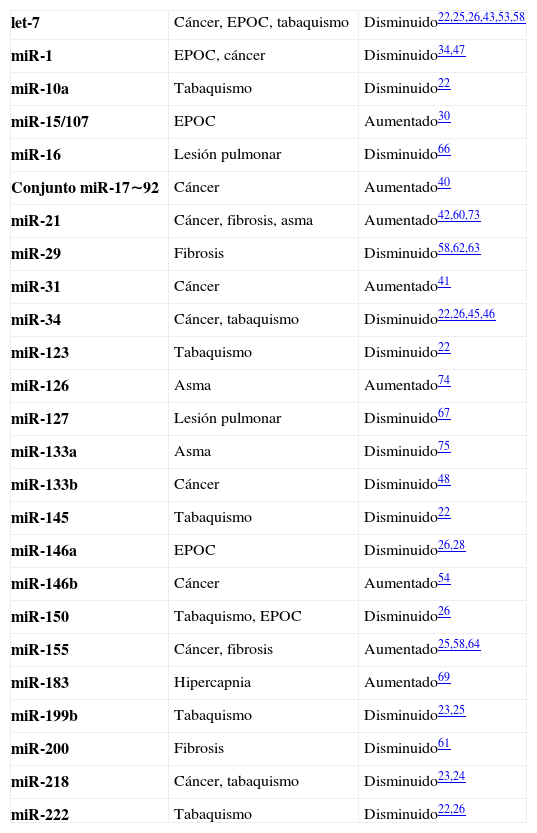
TabaquismoEl tabaquismo constituye el principal factor de riesgo para el desarrollo de enfermedad pulmonar obstructiva crónica (EPOC) y cáncer de pulmón y se estima sea responsable de casi 5 millones de muertes por año20. Se ha demostrado que el humo de tabaco provoca cambios en la expresión génica del epitelio respiratorio, vinculados al desarrollo de estas enfermedades21. Alteraciones en los niveles de ciertos miRs parecen estar involucrados en este proceso (tabla 1). Así, se han encontrado cambios significativos en la expresión pulmonar de 133 miRs en ratas expuestas a humo de cigarrillo, algunos de los cuales podrían participar del proceso carcinogénico22. Concretamente, favorecerían la proliferación celular y la angiogénesis, a la vez que disminuirían la actividad de mecanismos supresores tumorales. Entre los miRs cuya expresión está más disminuida se encuentran miR-34c y varios miembros de la familia let-7, lo que como veremos se ha vinculado a la patogenia del cáncer de pulmón. Junto con let-7, la reducción en los niveles de miR-10a, miR-123, miR-145 y miR-222 promueve la angiogénesis, fundamental para el desarrollo y crecimiento tumoral. Schembri et al. compararon el perfil de miRs a nivel del epitelio bronquial entre individuos fumadores y no fumadores, encontrando diferencias significativas en la expresión de 28 miRs23. Se destaca el hallazgo de niveles disminuidos de miR-199b y miR-218 en los fumadores, tal como se observa en pacientes con cáncer de pulmón24,25. Por su parte, Van Pottelberge y colaboradores reportaron diferencias en la expresión de 34 miRs a nivel del esputo inducido entre fumadores y no fumadores26. Entre ellos, la reducción de miR-150 podría tener relevancia en el deterioro de la función pulmonar vinculada al tabaquismo, ya que determinados genes implicados en la misma son diana de este miR. Integrantes de la familia let-7, miR-34c, miR-218 y miR-222 son algunos de los miRs que se encontraron descendidos en al menos dos estudios, resaltando la importancia que los mismos podrían tener en la patogenia de enfermedades relacionadas al tabaquismo como la EPOC o el cáncer de pulmón22,23,26.
MicroARNs en enfermedades pulmonares
| let-7 | Cáncer, EPOC, tabaquismo | Disminuido22,25,26,43,53,58 |
| miR-1 | EPOC, cáncer | Disminuido34,47 |
| miR-10a | Tabaquismo | Disminuido22 |
| miR-15/107 | EPOC | Aumentado30 |
| miR-16 | Lesión pulmonar | Disminuido66 |
| Conjunto miR-17∼92 | Cáncer | Aumentado40 |
| miR-21 | Cáncer, fibrosis, asma | Aumentado42,60,73 |
| miR-29 | Fibrosis | Disminuido58,62,63 |
| miR-31 | Cáncer | Aumentado41 |
| miR-34 | Cáncer, tabaquismo | Disminuido22,26,45,46 |
| miR-123 | Tabaquismo | Disminuido22 |
| miR-126 | Asma | Aumentado74 |
| miR-127 | Lesión pulmonar | Disminuido67 |
| miR-133a | Asma | Disminuido75 |
| miR-133b | Cáncer | Disminuido48 |
| miR-145 | Tabaquismo | Disminuido22 |
| miR-146a | EPOC | Disminuido26,28 |
| miR-146b | Cáncer | Aumentado54 |
| miR-150 | Tabaquismo, EPOC | Disminuido26 |
| miR-155 | Cáncer, fibrosis | Aumentado25,58,64 |
| miR-183 | Hipercapnia | Aumentado69 |
| miR-199b | Tabaquismo | Disminuido23,25 |
| miR-200 | Fibrosis | Disminuido61 |
| miR-218 | Cáncer, tabaquismo | Disminuido23,24 |
| miR-222 | Tabaquismo | Disminuido22,26 |
EPOC: enfermedad pulmonar obstructiva crónica.
La EPOC constituye la cuarta causa de muerte en el mundo, y se estima que constituya la tercera para el año 202027. Pese a que se ha aprendido mucho sobre la patogenia de esta enfermedad con componentes inflamatorios sistémicos, pocos estudios se han enfocado hasta el momento en el papel de los miRs en la EPOC (tabla 1). Sato et al. reportaron que los fibroblastos pulmonares de pacientes con EPOC presentan una menor expresión de miR-146a tras la estimulación con citoquinas proinflamatorias, en comparación con sujetos sin EPOC pero similar historia de tabaquismo28. Esto provoca una sobreexpresión de ciclooxigenasa-2 (diana de miR-146a) y subsecuente incremento de la producción de prostaglandina E2 (PGE2). Interesantemente, la producción de PGE2 y la expresión de miR-146a post-estimulación están asociados con la gravedad de la enfermedad explorada a través del volumen espiratorio forzado en el primer segundo (VEF1)28.
El análisis del perfil de miRs en el esputo inducido de pacientes con EPOC muestra diferencias con el de no fumadores o fumadores sin obstrucción al flujo aéreo26. En particular, la expresión de let-7c y miR-146a es menor en individuos con EPOC que continúan fumando que en fumadores sin obstrucción de la vía aérea. El receptor tipo II del factor de necrosis tumoral (RFNT-II), implicado en la patogenia de la EPOC, es uno de los ARNm diana de let-7c. En estos pacientes, la menor expresión de let-7c en el esputo se correlaciona inversamente con la concentración del RFNT-II. Existe además una correlación entre el nivel de let-7c y el VEF1. De esta forma, la disminución de miR-146a y let-7c podrían ser dos de los determinantes del estado inflamatorio y la progresión de la EPOC. Por otro lado, teniendo en cuenta que la reducción en los niveles de let-7 está vinculada a la carcinogénesis, surge la interrogante acerca de si alteraciones en la expresión de ciertos miRs pueden contribuir al riesgo aumentado de cáncer de pulmón en pacientes con EPOC26,29.
Recientemente, Ezzie et al. estudiaron los niveles de miRs en tejido pulmonar de fumadores con y sin EPOC30. Setenta miRs presentaron distinto grado de expresión entre ambos grupos. Algunas de estas alteraciones, como el incremento de integrantes de la familia miR-15/107, podrían afectar vías de señalización importantes en la patogenia de la EPOC, como la del factor transformador de crecimiento β (TGF-β) o las proteínas de la familia Wnt.
La disfunción muscular periférica es una manifestación frecuente en la EPOC, asociada a peor calidad de vida y mayor mortalidad31,32. La atrofia y el cambio en el tipo de fibra muscular (disminución del porcentaje de fibras de contracción lenta e incremento de fibras de contracción rápida) constituyen el fenotipo característico33. Lewis et al. encontraron una reducción de 2,5 veces en la expresión de miR-1 en el cuádriceps de pacientes con EPOC, en comparación con individuos sanos34. Los autores han demostrado también una correlación positiva entre los niveles de miR-1 e índices funcionales y de gravedad de la enfermedad como el VEF1, el test de marcha de 6 minutos y el cuestionario respiratorio St. George, así como con el porcentaje de fibras musculares lentas (tipo I). A su vez, los pacientes presentaron mayores niveles de la proteína histona deacetilasa 4 (cuyo ARNm es diana de miR-1) a nivel muscular, que podría explicar una vinculación entre el descenso de miR-1 y el cambio de tipo de fibra34. Otros miRs relevantes en la biología muscular, como miR-206, miR-208 o miR-499 no presentaron diferencias significativas entre pacientes con EPOC e individuos sanos34.
Cáncer de pulmónEl cáncer de pulmón representa una importante causa de mortalidad a nivel mundial35. A pesar de los avances en técnicas diagnósticas y terapéuticas, la supervivencia a 5 años sigue siendo muy baja36. Los miRs participan activamente en la regulación de procesos celulares tales como la proliferación, diferenciación y apoptosis. No resulta sorprendente por lo tanto la cantidad de evidencia sobre anormalidades en la expresión de miRs en distintos tipos de cáncer, actuando en ciertos casos como oncogenes (denominados oncomiRs) y en otros como supresores tumorales. El cáncer de pulmón, especialmente de células no pequeñas (CPCNP), es la patología respiratoria en la cual los miRs han sido estudiados más extensamente (tabla 1). Existe gran interés en la posible utilidad de los mismos en la detección temprana de la enfermedad, subclasificación de los distintos tipos de cáncer, estadificación pronóstica, identificación de pacientes con mayor probabilidad de resistencia a la quimioterapia y eventual herramienta terapéutica37,38.
La reducción en los niveles de la enzima Dicer, necesaria para la maduración de los miRs, ha sido reportada en el tejido tumoral de pacientes con cáncer de pulmón, en los que tuvo impacto pronóstico en la supervivencia39. Los miembros del conjunto miR-17∼92, sobreexpresados en el cáncer de pulmón de células pequeñas, favorecen el desarrollo tumoral y la neovascularización37,40. Liu et al. también demostraron el rol de miR-31 como oncomiR en el CPCNP, donde actúa reprimiendo a genes supresores de tumor41. Por su parte, miR-21 presenta niveles incrementados en cáncer pulmonar tanto de pacientes fumadores como no fumadores42. Este miR promueve el crecimiento celular e inhibe la apoptosis, favoreciendo el crecimiento tumoral y el desarrollo de metástasis38. Los integrantes de la familia de miRs let-7 están caracterizados como genes supresores tumorales, reprimiendo oncogenes tales como ras, myc y HMGA237. La expresión de let-7 se encuentra frecuentemente disminuida en el cáncer de pulmón, impactando negativamente en la supervivencia de los pacientes tratados quirúrgicamente43. La transcripción de miR-34 es directamente inducida por el supresor tumoral p53 en respuesta al daño del ADN, inhibiendo la proliferación celular inapropiada44. La expresión de este miR se encuentra disminuida en el tejido tumoral, asociándose a un mayor riesgo de recaída tras la resección quirúrgica45,46. Se ha reportado además una disminución en los niveles de miR-1 y miR-133b en el tejido tumoral47,48. Esto se asocia a un incremento de algunos de sus compuestos diana (MET, Pim-1, HDAC4, MCL-1, BCL2L2) involucrados en la carcinogénesis pulmonar5,47,48. La menor expresión de miR-218 en tumores de pacientes con CPCNP también ha llevado que sea planteado como un posible miR supresor tumoral24.
El estudio de los miRs puede convertirse en una herramienta diagnóstica importante a corto y mediano plazo. La clasificación histológica del cáncer pulmonar puede ser incompleta, especialmente en casos de tumores poco diferenciados, o cuando el material biópsico es escaso. La presencia de mutaciones y expresión de determinados biomarcadores sugieren la posible existencia de un mayor número de subcategorías con distintos pronósticos. Por lo tanto, la detección de determinados miRs puede ser de utilidad para entender e identificar los diferentes tipos de cáncer37. Por otro lado, más allá de su análisis a nivel del tejido tumoral, los miRs pueden ser detectados en muestras obtenidas en forma mínimamente invasiva49. En este sentido, se han comenzado a describir patrones de expresión de miRs en suero y esputo que podrían facilitar la detección precoz de pacientes con cáncer de pulmón50–52. De la misma forma, el hallazgo de ciertos miRs en el tejido tumoral tiene implicaciones pronósticas. Yanaihara et al. reportaron que la expresión elevada de miR-155 y disminuida de let-7a-2 se correlaciona con peor supervivencia en pacientes con adenocarcinoma pulmonar25. Yu et al. describieron como el perfil de expresión de 5 miRs se asocia al pronóstico en pacientes con CPCNP, con let-7a y miR-221 actuando como protectores, mientras que miR-137, miR-372 y miR-182* incrementan el riesgo de muerte53. En pacientes con carcinoma de células escamosas, el aumento de miR-146b en el tejido tumoral está asociado a peor supervivencia54. A nivel sérico, la detección de elevados niveles de miR-486 y miR-30d junto a bajos niveles de miR-1 y miR-499 se ha asociado también a peor pronóstico55.
Finalmente, existe gran expectativa sobre el rol que los miRs puedan tener en el desarrollo de estrategias terapéuticas en el cáncer de pulmón. La resistencia a la quimio y radioterapia constituye uno de los principales problemas actuales para el tratamiento. La expresión de algunos miRs ha sido implicada en los mecanismos de resistencia terapéutica en el cáncer de pulmón49. Este descubrimiento deja abierta no solo la posibilidad de orientar el tratamiento en base el perfil de miRs, sino también la eventual modulación de los mismos para incrementar la sensibilidad del tumor a la quimioterapia. El campo de la manipulación de los miRs in vivo se encuentra aún en desarrollo. No obstante, tanto la inhibición de oncomiRs específicos, como la restauración de la expresión de miRs supresores tumorales podrían constituir un avance importante en el tratamiento del cáncer de pulmón.
Fibrosis pulmonar idiopáticaLa fibrosis pulmonar idiopática (FPI) es una enfermedad crónica caracterizada por la fibrosis del intersticio pulmonar y deterioro progresivo de la función respiratoria. La causa es desconocida y las posibilidades terapéuticas limitadas56. La fibrosis y distorsión del parénquima pulmonar pueden ser producto de la activación de las células del epitelio alveolar, estimulando una transición epitelio-mesenquimal (TEM) con acumulación de fibroblastos y miofibroblastos secretores de matriz extracelular57. Pese a que aún son escasos los trabajos al respecto, está comenzando a cobrar relevancia el rol de los miRs en la patogenia de la FPI (tabla 1). Pandit et al. reportaron diferencias significativas en la expresión de 46 miRs entre pulmones de pacientes con FPI y sujetos control58. Entre estos, se destaca la disminución de let-7d, cuyos niveles en el epitelio alveolar se correlacionan con la capacidad vital forzada. La citoquina TGF-β reprime la transcripción de let-7d, determinando un incremento de HMGA2, una de las dianas del miR. Tanto TGF-β como HMGA2 han sido implicados en el desarrollo de la TEM en la fibrosis pulmonar59. La represión de let-7d también podría generar la sobreexpresión de otros compuestos vinculados al proceso fibrótico como el factor de crecimiento similar a la insulina 1 y su receptor58.
Los niveles de miR-21 se encuentran aumentados a nivel pulmonar en pacientes con FPI y este incremento se localiza principalmente en los fibroblastos/miofibroblastos60. La expresión de miR-21 es inducida por TGF-β, mientras que miR-21 reprime a Smad7 promoviendo la activación de fibroblastos mediada por TGF-β. De esta forma, miR-21 participa en un circuito de retroalimentación positiva con TGF-β favoreciendo el proceso fibrótico. Yang et al. reportaron recientemente que la expresión de miR-200 está disminuida en el pulmón de pacientes con FPI (miR-200a, miR-200c) y en un modelo animal de fibrosis pulmonar (miR-200a, miR-200b, miR-200c)61. Los autores demostraron que miR-200 inhibe la TEM inducida por TGF-β en células epiteliales alveolares, posiblemente mediante la represión de ciertos factores de transcripción involucrados en el proceso, como GATA3, ZEB1 o ZEB2. Sin embargo, el propio TGF-β regula negativamente la expresión de miR-200. Interesantemente, tanto el tratamiento con miR-200 como la inhibición de miR-21 reducen la fibrosis pulmonar experimental en ratones60,61.
La expresión pulmonar de miR-29 está reducida en ratones tras la inducción de fibrosis pulmonar, modificando la expresión de diversos genes asociados a la matriz extracelular62. En pacientes con FPI, los niveles pulmonares de miR-29 también se encuentran disminuidos58,63. La inhibición de miR-29 parece estar mediada nuevamente por TGF-β62. Finalmente, miR-155 se encontró elevado a nivel pulmonar en pacientes con FPI y animales con fibrosis experimental58,64. Este miR reprime al factor de crecimiento de queratinocitos en los fibroblastos, e incrementa la migración de los mismos64.
Lesión pulmonarLa lesión pulmonar aguda (LPA) y el síndrome de distrés respiratorio agudo (SDRA) se caracterizan por lesión e inflamación pulmonar, determinando edema, disminución de la distensibilidad y compromiso del intercambio gaseoso65. Alteraciones en la expresión de determinados miRs podrían participar en la regulación del proceso inflamatorio y la reparación tisular en la LPA/SDRA (tabla 1). Los niveles de miR-16 se encuentran reducidos en la LPA experimental inducida por lipopolisacárido (LPS)66. A su vez, el tratamiento con miR-16 disminuye la expresión de las citoquinas proinflamatorias interleuquina (IL) 6 y factor de necrosis tumoral α (FNT-α) en macrófagos tras la exposición a LPS. Recientemente, Xie et al. también reportaron una disminución en la expresión pulmonar de miR-127 en un modelo animal de LPA no infecciosa y en macrófagos alveolares expuestos a LPS67. El tratamiento con miR-127 no solamente disminuyó la producción de IL-1β, IL-6 y FNT-α en macrófagos expuestos a LPS, sino que in vivo redujo el grado de lesión en la LPA experimental. El control ejercido por miR-127 sobre la inflamación pulmonar estaría mediado a través de la represión del receptor CD64 de los macrófagos.
Una vez establecida la lesión, la reparación del epitelio alveolar es crucial para la recuperación de los pacientes con LPA/SDRA. Este proceso requiere de la proliferación y migración de las células epiteliales. Muchos pacientes con LPA/SDRA desarrollan hipercapnia debido al compromiso del intercambio gaseoso a nivel pulmonar, o de la estrategia ventilatoria empleada en caso de requerir asistencia ventilatoria mecánica (hipercapnia permisiva). Evidencia reciente sugiere que la elevación de la presión parcial de CO2 tiene efectos deletéreos en el pulmón, independientemente de los efectos sobre el pH68. La hipercapnia determina un incremento de miR-183, provocando disfunción mitocondrial al disminuir la expresión de la enzima isocitrato deshidrogenasa 2 (IDH2)69. La inducción de miR-183 por hipercapnia reduce la proliferación de fibroblastos pulmonares y células epiteliales alveolares69. La reparación del epitelio alveolar in vitro se encuentra afectada tanto por la hipercapnia como por miR-183, si bien ambos factores no han sido estudiados conjuntamente70,71.
AsmaEl asma es una enfermedad inflamatoria de la vía aérea, caracterizada por respuesta anormal de los linfocitos T CD4 tipo Th2 frente a determinados antígenos72. En distintos modelos de asma experimental se observa un incremento en la expresión pulmonar de miR-2173. Esto contribuiría al proceso inflamatorio en la vía aérea mediante la represión de IL-12, favoreciendo la respuesta linfocitaria tipo Th2. Los niveles de miR-126 también están aumentados en el asma experimental y su inhibición reprime la activación de linfocitos Th2, previniendo el desarrollo de hipersensibilidad en la vía aérea74. A nivel de las células del musculo liso, la disminución de miR-133a parece aumentar la hiperreactividad bronquial en un modelo animal de asma, mediante un incremento en la expresión de RhoA75. No obstante, pese a la evidencia proveniente de los modelos experimentales, Williams et al. no encontraron diferencias en la expresión de miRs en biopsias de la vía aérea entre sujetos sanos y asmáticos leves76. Serán necesarios nuevos trabajos para comprender mejor el rol de los miRs en la patogenia del asma.
ConclusionesSi bien se ha demostrado ampliamente la relación de a varios miRs con las enfermedades pulmonares, aún queda mucho por comprender el rol que cada miR desempeña en la patogenia de las mismas (tabla 1). El ritmo con el que aumenta el conocimiento sobre los mismos hace especular que el conocimiento adquirido sobre los miRs pueda ser trasladado a la clínica, donde podrían constituir poderosas herramientas diagnósticas y terapéuticas. No obstante, la complejidad del sistema de control en que se encuentran inmersos y el impacto que la modificación de un miR puede tener sobre decenas de ARNm hacen que su modulación farmacológica constituya un gran desafío.
Conflicto de interesesLos autores declaran no tener conflicto de intereses.
FinanciaciónEste trabajo fue financiado en parte por la US National Institutes of Health (Grants HL-85534 y HL-48129).
Los autores agradecen al Dr. Markus Queisser por sus valiosos comentarios y sugerencias.