La enfermedad pulmonar obstructiva crónica (EPOC) es una entidad de presentación heterogénea. Por ello, se han intentado perfilar diferentes fenotipos y endotipos, que permitirían un manejo más diferenciado. El objetivo del proyecto Biomarcadores en la EPOC (BIOMEPOC) es identificar biomarcadores sanguíneos útiles para tipificar mejor a los enfermos. Se analizarán datos clínicos y muestras sanguíneas en un grupo de pacientes y controles sanos. El proyecto constará de fases de prospección y de validación. Se realizarán determinaciones analíticas sanguíneas con técnicas convencionales y de diversas ciencias «ómicas» (transcriptómica, proteómica y metabolómica). Las primeras se realizarán orientadas por hipótesis, mientras que con las segundas se realizará una exploración sin dicho condicionante. Finalmente se realizará un análisis multinivel. En el momento actual se han reclutado 269 pacientes y 83 controles, y se está iniciando el procesamiento de muestras. Con los resultados obtenidos se espera identificar nuevos biomarcadores que, en solitario o combinados, permitan una mejor tipificación de los pacientes.
Chronic obstructive pulmonary disease (COPD) is an entity with a heterogeneous presentation. For this reason, attempts have been made to characterize different phenotypes and endotypes to enable a more individualized approach. The aim of the Biomarkers in COPD (BIOMEPOC) project is to identify useful biomarkers in blood to improve the characterization of patients. Clinical data and blood samples from a group of patients and healthy controls will be analyzed. The project will consist of an exploration phase and a validation phase. Analytical parameters in blood will be determined using standard techniques and certain ‘omics’ (transcriptomics, proteomics, and metabolomics). The former will be hypothesis-driven, whereas the latter will be exploratory. Finally, a multilevel analysis will be conducted. Currently, 269 patients and 83 controls have been recruited, and sample processing is beginning. Our hope is to use the results to identify new biomarkers that, alone or combined, will allow a better characterization of patients.
La enfermedad pulmonar obstructiva crónica (EPOC) es una entidad clínica caracterizada por un deterioro, en muchos casos progresivo, de la función pulmonar, con períodos intercurrentes de exacerbación de los síntomas1,2. Presenta una prevalencia muy elevada y conlleva elevados costes sanitarios y sociales2,3. La causa principal de la EPOC es el hábito tabáquico, aunque existen otros factores que pueden contribuir a su aparición2,4. Una característica relevante de esta entidad es su presentación muy heterogénea, ya que la clásica afectación pulmonar es variada y además suele ir acompañada de manifestaciones extrapulmonares y de diversas comorbilidades5,6. Entre las primeras destacan la afectación del estado nutricional y del sistema muscular esquelético, mientras que las segundas incluyen alteraciones cardiovasculares y endocrinas, así como el cáncer de pulmón. Con frecuencia, a estas comorbilidades se añade el impacto negativo del envejecimiento (ya que la enfermedad suele requerir una exposición prolongada al tóxico para manifestarse clínicamente), y en muchos casos un entorno social desfavorable (lo que hace que impacte sobre todo en segmentos frágiles de la sociedad). En paralelo a esta conceptualización como entidad compleja y heterogénea, se ha constatado que la variable que expresa la intensidad de la obstrucción al flujo aéreo (el FEV1), utilizada hasta hace poco como único parámetro para clasificar la gravedad de los enfermos, resulta insuficiente a la hora de establecer su pronóstico y delimitar un adecuado manejo clínico y tratamiento, ya que se omiten otros elementos de gran importancia en el perfil de la enfermedad.
Todo ello ha llevado en los últimos años a la búsqueda de perfiles más específicos en los enfermos, con el fin de ajustar de forma más individualizada su cuidado clínico, tratamiento y seguimiento continuado. En ese sentido, la clasificación denominada GOLD ha añadido recientemente el nivel de síntomas y número de exacerbaciones anuales al ya clásico FEV12. En un paso más, otros grupos han intentado definir perfiles con mayor especificidad, proponiendo el término «fenotipo», utilizado aquí de forma algo diferente de su semántica clásica, ya que haría referencia a un(os) rasgo(s) clínico(s) o biológico(s) con consecuencias en el pronóstico o el manejo terapéutico de los pacientes7. En este sentido, a los dos fenotipos clásicos de la enfermedad (enfisema pulmonar y bronquitis crónica), se han añadido recientemente clasificaciones que incluyen, entre otros, los fenotipos «exacerbador frecuente» y «mixto» (p.ej., de EPOC-asma o ACO, por asthma-COPD overlap)8,9. Otros grupos han descrito diversos fenotipos basados en algún rasgo clínico relevante (p.ej., la pérdida de peso, la comorbilidad cardiovascular o la persistencia de inflamación sistémica)10-13. También se ha intentado perfilar una caracterización basada en los mecanismos fisiopatológicos que explican la heterogeneidad (endotipos), aunque esta visión es todavía más incipiente. Más recientemente, se ha propuesto dar un paso más, aproximándose a la caracterización individualizada de los pacientes13. En este enfoque de medicina personalizada se hace necesario disponer de suficientes indicadores que permitan una filiación lo más exacta posible de cada enfermo. No solo es necesario definir la gravedad de la enfermedad pulmonar (hasta ahora basada en la reserva funcional), sino otras dimensiones como su grado de actividad (intensidad de los mecanismos biológicos que caracterizarían a los endotipos) o el impacto que supone en la vida del paciente14, y tanto sus comorbilidades como sus manifestaciones sistémicas.
Este cambio conceptual implica a nivel práctico la búsqueda de nuevas variables (marcadores) que, idealmente, sean fáciles de obtener y permitan una adecuada clasificación de los pacientes. La búsqueda de dichas variables debería responder a las características heterogéneas y complejas de la enfermedad, y basarse en la interrelación de los diversos niveles que condicionan su expresión. Se trata pues de abordar la complejidad de la enfermedad, permitiendo una estratificación apropiada y ligada a determinadas características biológicas15. Si hasta ahora se ha trabajado fundamentalmente con los niveles ambientales y clínicos, estos deben ser enriquecidos con los diferentes niveles biológicos, para obtener los denominados endotipos. Esto solo será posible mediante la identificación de biomarcadores, definidos como indicadores objetivos de un estado biológico15, habitualmente detectables en alguno de los fluidos biológicos o tejidos16.
Lamentablemente, hasta la fecha los intentos para obtener buenos marcadores biológicos no han dado en la EPOC los resultados apetecidos. Por un lado, existen numerosos trabajos que han explorado un número reducido de moléculas, estando en general orientados a una hipótesis basada en los conocimientos fisiopatológicos actuales (inflamación, estrés oxidativo, lesión o remodelación tisular)17-19. Suele tratarse de estudios con series relativamente amplias de pacientes, orientados a una circunstancia clínica concreta (p.ej., frecuencia de exacerbaciones, rapidez evolutiva), y en muchos casos sin fase de validación posterior. En general los resultados han sido poco concluyentes, aunque el estudio ECLIPSE permitió observar que la combinación de diversos marcadores inflamatorios séricos puede mejorar la predicción evolutiva de las variables clínicas clásicas17. También en los últimos años y con objetivos similares, se han realizado diversos estudios utilizando las llamadas ciencias y técnicas «ómicas». Estas se caracterizan en general por una búsqueda amplia, sin hipótesis concreta preestablecida. Sin embargo, en general se han empleado series relativamente cortas o escasamente filiadas desde un punto de vista clínico, por lo que tampoco se han alcanzado los resultados apetecidos20-25. El presente proyecto pretende superar algunas de las mencionadas limitaciones.
La hipótesis general del proyecto Biomarcadores en la EPOC (BIOMEPOC) es que la búsqueda de biomarcadores de las diferentes circunstancias clínicas características de la EPOC (eventos agudos intercurrentes, grado de actividad, presencia de eosinofilia, comorbilidades, manifestaciones sistémicas, etc.) será mucho más eficiente empleando conjuntamente metodologías clásicas y de biología de sistemas. Esta aproximación permitirá definir redes de interacción, sondeando vías metabólicas y eventuales mecanismos moleculares involucrados en cada caso, identificando en último término los biomarcadores más adecuados. Lógicamente, la hipótesis específica del abordaje con técnicas «ómicas» derivará de la prospección inicial y de las moléculas allí identificadas, que serán validadas posteriormente. Por el contrario, la hipótesis específica de la parte del estudio que utiliza técnicas convencionales es que los marcadores inflamatorios, de estrés oxidativo y de lesión-reparación en los tejidos pulmonar, muscular y cardiovascular pemitirán distinguir las diferentes formas, fenotipos y endotipos ligados a manifestaciones pulmonares y sistémicas de la enfermedad. Consecuentemente, el objetivo general del proyecto es analizar de forma integrada los indicadores obtenidos a partir de la clínica, las determinaciones analíticas convencionales y los datos biológicos masivos y multinivel en pacientes con EPOC, con el fin de definir nuevos perfiles específicos o fenotipar y endotipar mejor algunos de los ya propuestos.
De las cuatro modalidades de biomarcadores definidas por la Comisión Europea (Susceptibilidad/Riesgo, Diagnóstico, Pronóstico y Predicción de tratamiento)15, el proyecto BIOMEPOC, de diseño inicialmente transversal, se centra en las dos primeras. Sin embargo, estará entre sus objetivos futuros el cumplir también con la tercera y cuarta modalidades, aunque ello implica un seguimiento más prolongado que excede a la duración inicial del estudio.
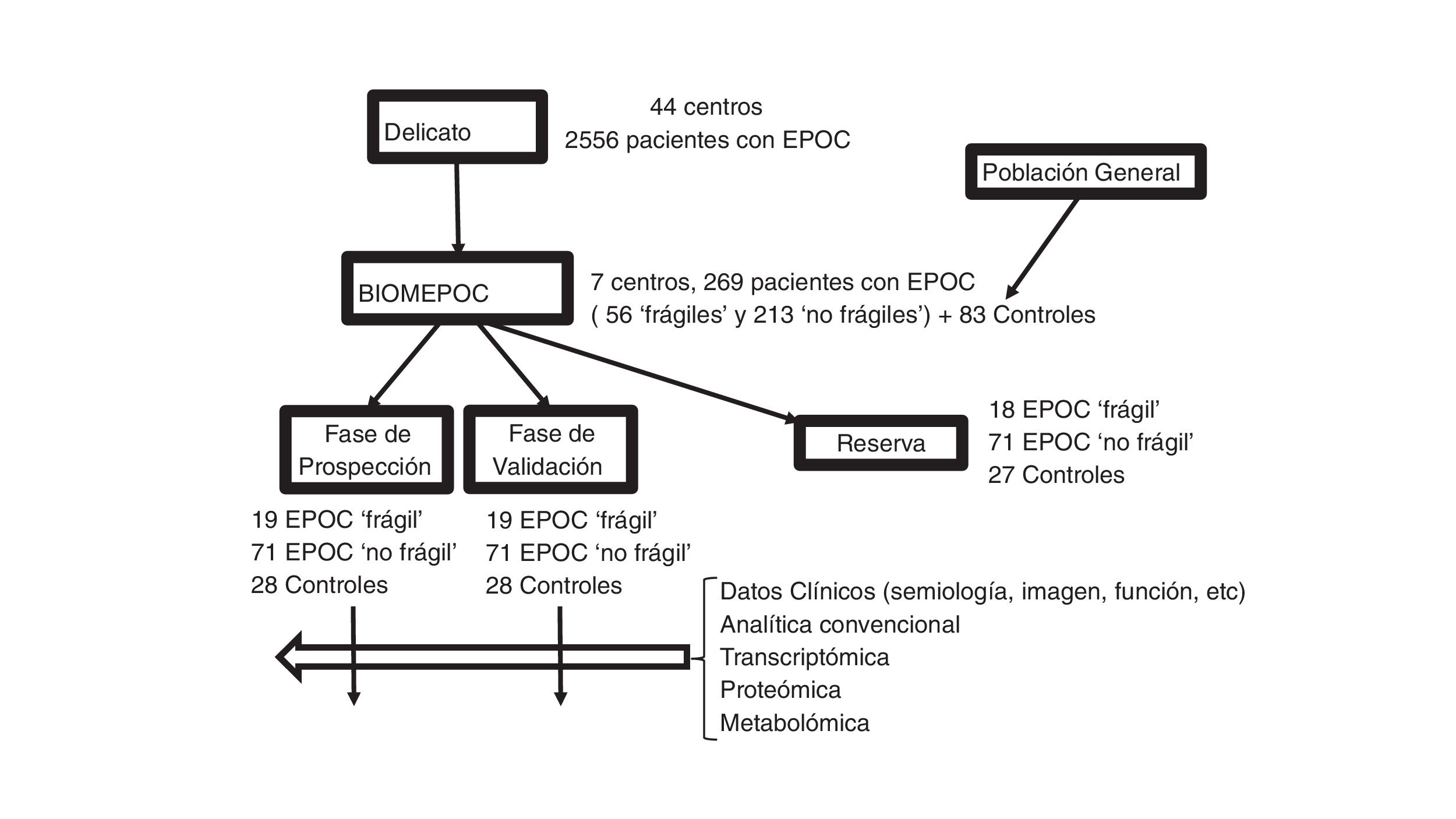
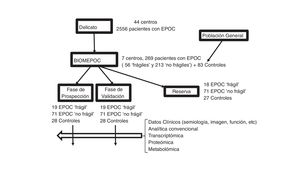
MétodoDiseño, población y variables clínicasEl estudio BIOMEPOC es de tipo prospectivo (que incluye la adquisición de numerosas variables y muestras biológicas en el momento del reclutamiento, pero también el seguimiento de algunas variables como la aparición posterior de cáncer de pulmón y mortalidad), con inclusión de pacientes procedentes en su mayor parte de un estudio previo y de controles sanos en los 7 centros participantes (Hospital Son Espases de Palma, Fundación Jiménez Díaz de Madrid, Hospital Clínic de Barcelona, Hospital Universitario Virgen del Rocío de Sevilla, Hospital 12 de Octubre de Madrid, Hospital del Mar de Barcelona y Hospital Universitari Parc Taulí de Sabadell). Se ha escogido un perfil de hospital terciario y universitario, acostumbrado tanto al manejo de pacientes complejos como de muestras biológicas que requieren manejo especializado. El proyecto consta de dos fases. La primera de ellas, de prospección y selección de biomarcadores, utilizando un subgrupo de pacientes y de controles sanos; y una segunda fase, de validación interna de los resultados ya obtenidos utilizando otro subgrupo de pacientes y de controles sanos (fig. 1).
El planteamiento del proyecto no sería posible sin la disponibilidad de un grupo multicéntrico con amplia y larga experiencia en trabajos colaborativos realizados en red, así como de una cohorte de pacientes bien tipificados clínicamente. Afortunadamente existe un estudio de auditoría clínica denominado «Design and local implementation of Clinical Audits in different types of Obstructive Lung Disease» (DELICATO), promovido conjuntamente por el Área de Respiratorio del Centro de investigación en Red (CIBERES) y la Sociedad Española de Neumología y Cirugía Torácica (SEPAR), que incluye una cohorte apropiada para los objetivos del presente proyecto, y a la que se nos dio acceso.
Por tanto, los criterios de inclusión y exclusión de los pacientes fueron los mismos que en DELICATO. De forma breve, se estudiaron pacientes con EPOC2 y seguimiento continuado en una consulta especializada de neumología de al menos 2 años, a los que se añadieron controles sanos (seleccionados mediante cuestionario estandarizado y pruebas de función respiratoria dentro de la normalidad; con sexo y edad equilibrados con los de los pacientes), todos ellos mayores de 40 años. Los criterios diagnósticos para la EPOC fueron historia de hábito tabáquico, así como un cociente espirométrico porcentual posterior a la prueba broncodilatadora inferior al 70%2. El cálculo del tamaño muestral se realizó a partir de estudios previos18,26-28, tomándose el de mayor envergadura26 para el cálculo del número total de pacientes con EPOC, y ampliándolo generosamente en previsión de las pérdidas o dificultades de reclutamiento. Los otros estudios se utilizaron para el cálculo de muestras sanguíneas a valorar en los subanálisis empleando las distintas técnicas ya mencionadas. Esto supuso que cada uno de los 7 centros debía inicialmente intentar reclutar 90 pacientes con EPOC (unos 30 pacientes considerados «frágiles» según la definición adoptada en DELICATO: 3 o más contactos hospitalarios por exacerbación en el año precedente; y 60 enfermos más sin dicha condición) y 30 sujetos sanos. En todos ellos se extraería una muestra sanguínea para el estudio de biomarcadores, así como muestra espontánea de esputo para ulteriores estudios en aquellos pacientes en que esto fuese posible.
Aspectos éticosEl estudio se diseñó atendiendo a los principios recogidos en la declaración de Helsinki, siendo aprobado por los Comités de Ética e Investigación Clínica (CEIC) de las diferentes instituciones. Todos los participantes en el estudio firmaron el correspondiente consentimiento informado tras haberles sido detallados los objetivos y procedimientos a emplear. Para garantizar el anonimato, se asignó a cada participante y a sus muestras un número correlativo de 4 cifras. El coordinador general del proyecto mantiene en archivo aparte y seguro la correspondencia entre dichas cifras y la identidad del sujeto.
Datos clínicosSe recogió en una nueva base de datos centralizada, actualizándola, la información clínica, analítica, funcional y de seguimiento obtenida en el proyecto DELICATO. La presencia y severidad del enfisema se valoró a partir de la radiología convencional de tórax y la medición de la transferencia del CO, mientras que el estado nutricional se evaluó a través del valor del índice de masa corporal. A la mencionada base de datos se añadieron los correspondientes a los controles sanos.
Análisis de biomarcadoresPara la obtención de los biomarcadores se han utilizado diversas estrategias complementarias. En primer lugar, y para las técnicas convencionales en las que debían definirse previamente las moléculas a analizar, estas fueron seleccionadas a partir de la literatura especializada y un trabajo ya publicado por parte del grupo investigador utilizando la «minería de textos» (text mining)29. Por el contrario, y como ya se ha mencionado, no se estableció ninguna selección para las técnicas «ómicas» de screening amplio, evitando el potencial sesgo de una hipótesis predefinida.
Procesamiento y análisis de las muestras sanguíneasPrevio al inicio del proyecto se elaboró y distribuyó entre los participantes el protocolo estandarizado de recogida y procesamiento inicial de muestras (addendum 1). Estas fueron remitidas al centro coordinador y almacenadas a −80°C en su Biobanco (MARBiobanc). Dicha instalación cumple los criterios estandarizados de seguridad y calidad para la preservación y custodia de muestras biológicas (AENOR ER-0031/2012; UNE-EN ISO 90019). Las muestras serán procesadas con técnicas analíticas convencionales y análisis amplio de los niveles transcriptómico, proteómico y metabolómico. Se reserva una parte de las muestras por si fuera necesario utilizarlas en ulteriores análisis.
- Técnicas «convencionales». Incluye hemograma completo y determinaciones séricas de analítica convencional y de marcadores genéricos de inflamación [proteína C reactiva (CRP) ultrasensible, fibrinógeno y glico-marcadores], estrés oxidativo [8-isoprostano, malondialdehído, glutatión reducido, proteína secretora de las células «club» (CC) -16 y granzima (Gzm) B], infección (procalcitonina) y disfunción cardiovascular [péptido natriurético tipo B (BNP) y pro-BNP].
- Análisis transcriptómico. Se utilizan muestras tratadas de forma específica para preservar el ácido ribonucleico (ARN). Estas muestras se procesan para secuenciación masiva (NextSeq, Illumina, San Diego, EE.UU.), según procedimiento ya descrito en la literatura30, lo que permite el análisis de más de 25.000 genes por muestra. Dicho análisis se realiza en la plataforma de transcriptómica de la Universidad Pompeu Fabra de Barcelona (UPF). Los resultados son analizados por bioinformáticos y clínicos de nuestro grupo mediante programas específicos convencionales, con análisis funcional posterior a través de Gene Ontology (GO, Gene Ontology Consortium)31 y Human Phenotype Ontology (HPO, Charité, Berlín, Alemania)32. Los resultados de transcriptomas seleccionados entre los que demuestren un valor discriminatorio elevado se confirmarán por la técnica cuantitativa de reacción en cadena de la polimerasa con transcriptasa inversa (qRT-PCR).
- Análisis proteómico. Incluye tanto técnicas masivas (para proteínas de mediana/alta concentración sanguínea) como semimasivas (para proteínas con concentración más baja). Para las primeras se empleará la espectrometría de masas tríbrida de alta velocidad (Orbitrap Fusion Lumos, Filgen Inc., Nagoya, Japón), a realizar en el Centro de Regulación Genómica (CRG) de Barcelona según metodología estándar33, mientras que las segundas se basan en arrays (Affimetrix) dirigidos a facetas biológicas concretas, seleccionadas por los procedimientos ya mencionados. En concreto, y siguiendo una técnica convencional34, se estudiarán 45 biomarcadores-diana, incluyendo hormonas y moléculas relacionadas [hormonas adrenocorticotropa (ACTH), del crecimiento (GH), y estimulante del tiroides (TSH), péptido C, ghrelina, glucagón, insulina y leptina], marcadores de inflamación [quimioquina expresada y secretada por células T, regulada por activación (RANTES), interferón (IFN) -α2, IFN-γ, interleucina (IL) -1α, IL-1β, IL-1ra, IL-2, IL-3, IL-4, IL-5, IL-6, IL-7, IL-8, IL-10, IL-12 (p40), IL-12 (p70), IL-13, IL-15, IL-17, IP-10, proteína quimiotáctica del monocito (MCP) -1, proteína inflamatoria del macrófago (MIP) -1α, MIP-1β, factor de necrosis tumoral (TNF) -α, TNF-β, factor de crecimiento transformante (TGF) -β y factor de crecimiento endotelial vascular (VEGF)], marcadores de lesión o remodelado tisular (troponina C, desmosina, elastina y proteína D del surfactante), de infección [procalcitonina y proteína ligada al polisacárido (LBP)] y de disfunción endotelial [molécula soluble de adhesión intercelular (sICAM) -1]. Con posterioridad se determinarán mediante análisis por inmunoabsorción ligado a enzimas (ELISA) los marcadores seleccionados por mostrar señal altamente discriminatoria con las técnicas de rastreo anteriores.
- Análisis metabolómico. Se efectuará mediante espectrometría de masas con analizadores tipo quadrupolo-tiempo de vuelo (Q-TOF-MS) (QSTAR XL Hybrid System, Applied Biosystems. Foster City, CA, EE.UU.), y fuentes de ionización tipo electrospray (ESI) o fotospray (APPI). La muestra se introducirá por infusión directa (DIMS) mediante bomba de baja presión y jeringa tipo Hamilton de 1000μL, a un flujo de 5μL/min. El estudio se completará con el realizado mediante cromatografía de gases (GC-MS), orientado a los metabolitos más volátiles. En los casos en que se estime necesario se obtendrán perfiles metabólicos adicionales mediante espectrometría de masas por cromatografía líquida de ultra alta resolución, también con analizadores tipo quadrupolo-tiempo de vuelo (UHPLC-Q-TOF-MS). En todos los casos se utilizará metodología ya validada en la literatura35. Para el análisis de resultados se emplearán las bases de datos metabólicas METLIN Metabolomics Database (Scripps Research Institute, San Diego, CA, EE.UU.), FIEHN (NIH West Coast Metabolomics Center, Davis, CA, EE.UU.), HMDB (Human Metabolome Database, TMIC, Edmonton, AL, Canadá) y AMIX (Bruker, Billerica, MAS, EE.UU.), entre otras.
Análisis de datos y procesamiento estadísticoLos datos clínicos, funcionales y biológicos convencionales se han recogido en una base de datos única. En cuanto al análisis de los niveles transcriptómico, proteómico y metabolómico, con evaluación posterior de sus interacciones recíprocas, se emplearán las plataformas y paquetes estadísticos antes citados, así como los programas SPSS 18.0 (IBM Corp.) y R (GPL, programa de uso libre), mediante paquetes de software de BIOCONDUCTOR (PMID:25633503). Se establecerán comparaciones atendiendo a diferentes criterios dicotómicos, que incluyen presencia de enfermedad, gravedad de esta según la alteración funcional (leves-moderados frente a graves-muy graves), presencia o no de «fragilidad» (definición ya mencionada), presencia o no de eosinofilia, presencia o no de alteraciones nutricionales, presencia o no de enfisema marcado, presencia o no de bronquiectasias, sexo del paciente, etc. Para el control de calidad y evaluación de los posibles efectos de homogeneidad por grupo de muestras (batch) se utilizará el módulo de componentes principales de Partek. Las relaciones entre variables se calcularán mediante coeficientes convencional y biserial de Pearson. También se realizará un análisis multivariado de regresión logística, que permitirá ajustar el efecto de las distintas covariables. Para ello se diseñará un modelo normal multivariable (MVN) mediante el paquete R «nlme» (función «gls»), lo que permitirá valorar asociaciones entre los diferentes niveles clínicos y biológicos. Estos modelos consideran todos los perfiles ómicos simultáneamente como un resultado multivariado Y. La matriz de varianza-covarianza se modela por tanto de manera flexible, por lo que la covarianza depende de las variables clínicas principales y permite diferentes coeficientes para cada condición clínica escogida. Así, la variable biológica escogida (Y) para cada individuo y escenario clínico (p.ej., su fenotipo) sigue una distribución normal multivariada con un vector promedio (μ) y una matriz de covarianza (Σ) Y ∼ MVN(μ, Σ). La matriz de varianza-covarianza no estructurada se modela utilizando la identificación de los sujetos como factor de agrupamiento. La integración de datos de las diversas «ómicas» se llevará a cabo mediante el análisis de correlaciones canónicas múltiples (PMID:19377034). Finalmente se realizará con los resultados un análisis de clusters para definir eventuales nuevos endotipos de la enfermedad, según metodología ya utilizada previamente por diversos autores del grupo10. Para el análisis se cuenta con la estructura humana y tecnológica del Programa de Investigación en Informática Biomédica [GRIB, perteneciente al Instituto Hospital del Mar de Investigación Médica (IMIM) y a la UPF].
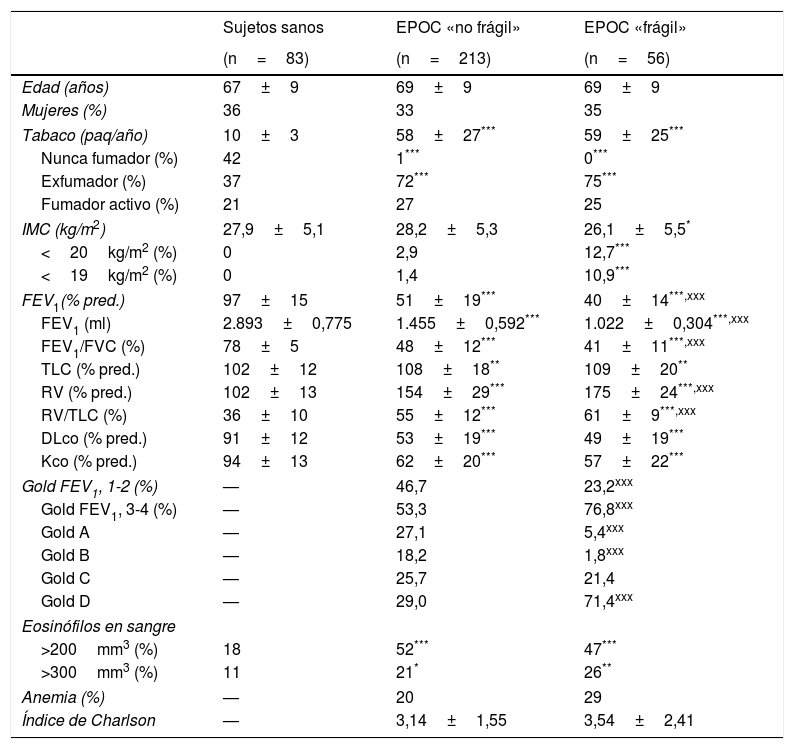
Descripción de la poblaciónEn el momento actual, se ha finalizado la fase de reclutamiento, que finalmente ha incluido 83 sujetos sanos [con sendos subgrupos de fumadores (56%) y de no fumadores], 213 pacientes con EPOC pero sin criterios de «fragilidad» y 56 pacientes más con EPOC y «fragilidad» (fig. 1). La reducción en el número de pacientes con EPOC se debió fundamentalmente a la dificultad de retomar a los pacientes del estudio DELICATO, tanto por fallecimientos como por cambios de domicilio o rechazo a participar en el presente estudio. No obstante, el número final de pacientes estables se aproximaba al 80% del inicialmente previsto. Se descartaron posteriormente 5 controles y 36 pacientes adicionales por no hallarse todas las muestras sanguíneas en excelentes condiciones. Fundamentalmente, esto fue debido a mala preservación de la muestra para análisis del transcriptoma. De todos los demás se dispone ya de los datos clínicos, procedentes en su mayoría del estudio de DELICATO, con actualización en el momento de la inclusión en el presente estudio (tabla 1). Se trata de pacientes con predominio del sexo masculino, buen estado nutricional salvo en el grupo de fragilidad (11-13% de bajo peso), con las alteraciones funcionales características de su enfermedad pulmonar. Atendiendo a su gravedad clasificada por el FEV1, los pacientes sin fragilidad se distribuían a partes iguales entre leves-moderados y graves-muy graves2, mientras que en los frágiles predominaban netamente los segundos. En cuanto a la clasificación GOLD más reciente, los pacientes sin fragilidad mostraban una distribución similar en las 4 categorías2, y en cambio los enfermos frágiles se concentraban en la D. Finalmente, ambos grupo de pacientes mostraban unos porcentajes similares de eosinofilia en sangre periférica, anemia y comorbilidades (índice de Charlson).
Características generales de la población incluida en el estudio BIOMEPOC
| Sujetos sanos | EPOC «no frágil» | EPOC «frágil» | |
|---|---|---|---|
| (n=83) | (n=213) | (n=56) | |
| Edad (años) | 67±9 | 69±9 | 69±9 |
| Mujeres (%) | 36 | 33 | 35 |
| Tabaco (paq/año) | 10±3 | 58±27*** | 59±25*** |
| Nunca fumador (%) | 42 | 1*** | 0*** |
| Exfumador (%) | 37 | 72*** | 75*** |
| Fumador activo (%) | 21 | 27 | 25 |
| IMC (kg/m2) | 27,9±5,1 | 28,2±5,3 | 26,1±5,5* |
| <20kg/m2 (%) | 0 | 2,9 | 12,7*** |
| <19kg/m2 (%) | 0 | 1,4 | 10,9*** |
| FEV1(% pred.) | 97±15 | 51±19*** | 40±14***,xxx |
| FEV1 (ml) | 2.893±0,775 | 1.455±0,592*** | 1.022±0,304***,xxx |
| FEV1/FVC (%) | 78±5 | 48±12*** | 41±11***,xxx |
| TLC (% pred.) | 102±12 | 108±18** | 109±20** |
| RV (% pred.) | 102±13 | 154±29*** | 175±24***,xxx |
| RV/TLC (%) | 36±10 | 55±12*** | 61±9***,xxx |
| DLco (% pred.) | 91±12 | 53±19*** | 49±19*** |
| Kco (% pred.) | 94±13 | 62±20*** | 57±22*** |
| Gold FEV1, 1-2 (%) | — | 46,7 | 23,2xxx |
| Gold FEV1, 3-4 (%) | — | 53,3 | 76,8xxx |
| Gold A | — | 27,1 | 5,4xxx |
| Gold B | — | 18,2 | 1,8xxx |
| Gold C | — | 25,7 | 21,4 |
| Gold D | — | 29,0 | 71,4xxx |
| Eosinófilos en sangre | |||
| >200mm3 (%) | 18 | 52*** | 47*** |
| >300mm3 (%) | 11 | 21* | 26** |
| Anemia (%) | — | 20 | 29 |
| Índice de Charlson | — | 3,14±1,55 | 3,54±2,41 |
% pred.: porcentaje sobre el predicho; DLco: factor de tansferencia para el CO; FEV1: volumen espiratorio forzado obtenido en el primer segundo; FVC: capacidad vital forzada; IMC: índice de masa corporal; Kco: índice de Krogh (transferencia de monóxido de carbono dividida por el volumen alveolar, DLco/VA); RV: volumen residual; TLC: capacidad pulmonar total.
Anemia, según valor de hemoglobina: ♂ y ♀ posmenopáusicas, ≤13g/L; ♀ premenopáusicas, ≤12g/L.
Comparaciones: Grupo de pacientes con Grupo control de sujetos sanos: *** p<0,001; ** p<0,01; * p<0,05; ambos grupos de pacientes entre sí: xxx p<0,001.
Por otra parte, las muestras biológicas fueron extraídas y sometidas a un procesamiento inicial. El proyecto BIOMEPOC se halla en fase de evaluación de los primeros resultados del nivel transcriptómico, habiéndose diseminado ya algunos resultados muy preliminares en forma de comunicaciones a congresos36-38.
Como se ha mencionado, el proyecto incluye diversos aspectos novedosos en la búsqueda de biomarcadores. Por un lado, ha utilizado la minería de textos para la generación de nuevas hipótesis, mientras que por otro utiliza técnicas de análisis semimasivo y masivo para ampliar el cribado a potenciales marcadores no regidos por hipótesis. Otro elemento destacable será el no limitarse al análisis de los resultados de cada técnica «ómica» por separado, sino integrarlos mediante redes de interacción que permitan relacionar dichos niveles entre sí (análisis multinivel). La complejidad de la enfermedad podrá ser explorada así desde una nueva óptica, complementada con los datos clínicos, funcionales y analíticos convencionales ya disponibles (a menudo ausentes en los estudios de biología más básica).
En resumen y como conclusión, el presente proyecto BIOMEPOC es uno de los primeros intentos para explorar de forma muy amplia los potenciales biomarcadores de diferentes aspectos relacionados con la heterogeneidad clínica que caracteriza a la EPOC. Su objetivo principal es llegar a identificar perfiles biológicos de pacientes (endotipos a un nivel elevado de complejidad) en función de determinados correlatos clínicos (fenotipos, descritos previamente o no). Potencialmente, también puede permitir explorar nuevos caminos terapéuticos para alguno de estos perfiles. Las principales novedades del proyecto son el reunir en un solo estudio un número suficiente de pacientes (sobre todo en el contexto de algunos análisis «ómicos»), disponer de una evaluación clínico-funcional completa realizada por especialistas en aparato respiratorio, y la utilización complementaria de técnicas convencionales y masivas de detección, con exploración simultánea de diversos niveles «ómicos» (transcriptomas, proteínas y metabolitos) y sus potenciales interacciones. Se espera generar así nuevo conocimiento desde una óptica multidisciplinar, con un propósito netamente de transferencia, tanto hacia el sistema de salud como eventualmente a la industria.
FinanciaciónFinanciado parcialmente por SAF2014-54371 (Fondos FEDER), SEPAR 2015 y 2016, FUCAP 2014 y Menarini 2015.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Mireia Admetlló (Hospital del Mar, CIBERES, Barcelona)
Alvar Agustí (Hospital Clínic, Universitat de Barcelona, CIBERES, Barcelona)
Carlos Alvarez-Martínez (Hospital 12 de Octubre, CIBERES, Madrid)
Esther Barreiro (Hospital del Mar, IMIM, Universitat Pompeu Fabra, CIBERES, Barcelona)
Carme Casadevall (IMIM, Universitat Pompeu Fabra, CIBERES, Barcelona)
Ferran Casals (Universitat Pompeu Fabra, Barcelona)
Robert Castelo (Universitat Pompeu Fabra, Barcelona)
Ady Castro-Acosta (Hospital 12 de Octubre, CIBERES, Madrid)
Rocío Córdova (Hospital Son Espases, CIBERES, Palma)
Borja G. Cosío (Hospital Son Espases - Instituto de Investigación Sanitaria de Baleares [IdISBa], CIBERES, Palma de Mallorca)
Rosa Faner (Fundació Clínic per la Recerca Biomédica, CIBERES, Barcelona)
Laura I. Furlong (IMIM, Barcelona)
Marian García (Consorci Hospitalari de Parc Taulí, Universitat Autònoma de Barcelona, CIBERES, Sabadell)
Joaquim Gea (Hospital del Mar, IMIM, Universitat Pompeu Fabra, CIBERES, Barcelona)
José G. González-García (Hospital del Mar, Barcelona)
Carmen Hernández-Carcereny (Hospital Clínic, Universitat de Barcelona, CIBERES, Barcelona)
José Luis López-Campos (Hospital Universitario Virgen del Rocío, Universidad de Sevilla, CIBERES)
Eduardo Márquez (Hospital Universitario Virgen del Rocío, Universidad de Sevilla, CIBERES)
Eduard Monsó (Consorci Sanitari Parc Taulí, Universitat Autònoma de Barcelona, CIBERES, Sabadell)
Concepción Montón (Consorci Sanitari Parc Taulí, Universitat Autònoma de Barcelona, CIBERES, Sabadell)
Miren Josune Ormaza (Consorci Sanitari Parc Taulí, Universitat Autònoma de Barcelona, CIBERES, Sabadell)
Alexandre Palou (Hospital Son Espases, CIBERES, Palma de Mallorca)
Sergi Pascual (Hospital del Mar, IMIM, Universitat Pompeu Fabra, CIBERES, Barcelona)
Germán Peces-Barba (Fundación Jiménez Díaz, Universidad Autónoma de Madrid, CIBERES, Madrid)
Pau Puigdevall (Universitat Pompeu Fabra, Barcelona)
Ferran Sanz (IMIM, Universitat Pompeu Fabra, Barcelona)
Luis Seijó (Fundación Jiménez Díaz, Universidad Autónoma de Madrid y Universidad de Navarra, CIBERES, Madrid)
Montserrat Torà (IMIM, Universidad Autónoma de Barcelona, Barcelona)
Yolanda Torralba (Hospital Clínic, CIBERES, Barcelona)
Carles Vilaplana (Laboratorio de Referencia de Catalunya, El Prat de Llobregat).