Cada vez es más habitual disponer de información médica en formato electrónico. Esto incluye tanto artículos científicos como revisiones sobre el manejo clínico e incluso registros de instituciones sanitarias con datos de pacientes. Sin embargo, los instrumentos tradicionales, tanto individuales como institucionales, son poco útiles para seleccionar la información más apropiada en cada caso, sea en el ámbito clínico o en el de la investigación. La llamada «minería» de textos o de datos permite gestionar esa gran cantidad de información, extrayéndola de fuentes diversas mediante sistemas de procesamiento (filtrado y curado), integrándola y permitiendo la generación de nuevo conocimiento. La presente revisión pretende proporcionar una idea general sobre la minería de textos y datos, así como sobre la ayuda que esta técnica bioinformática puede suponer para el ejercicio asistencial de la medicina respiratoria y para la investigación en ese mismo campo.
It is increasingly common to have medical information in electronic format. This includes scientific articles as well as clinical management reviews, and even records from health institutions with patient data. However, traditional instruments, both individual and institutional, are of little use for selecting the most appropriate information in each case, either in the clinical or research field. So-called text or data «mining» enables this huge amount of information to be managed, extracting it from various sources using processing systems (filtration and curation), integrating it and permitting the generation of new knowledge. This review aims to provide an overview of text and data mining, and of the potential usefulness of this bioinformatic technique in the exercise of care in respiratory medicine and in research in the same field.
Las enfermedades respiratorias más prevalentes (asma bronquial, enfermedad pulmonar obstructiva crónica [EPOC], infecciones y cáncer de pulmón) suponen un gran reto para la salud y para los sistemas sanitarios, dado el elevado coste económico y social que representan1–7. Por otra parte, con el avance de las tecnologías relacionadas con la información es posible acceder y analizar cantidades ingentes de datos relacionados con la salud y la enfermedad. Así, es común registrar datos de pacientes en formato electrónico, tanto en los hospitales como en la medicina primaria. Esto incluye datos de filiación, pero también los relacionados con los diagnósticos, la gravedad, los resultados analíticos, las pruebas funcionales y la medicación, así como las características de los contactos del enfermo con el sistema sanitario. Disponer de esta gran cantidad de información en formato digital ofrece 3ventajas importantes: a)mejora su calidad; b)reduce el tiempo marginal de trabajo por parte del personal sanitario, y c)ofrece la posibilidad de utilizar dicha información mediante sistemas automatizados, como la «minería de textos» (text mining) o la «minería de datos» (data mining)8,9. Desde un punto de vista estricto, se diferencian en que la primera obtiene la información a partir de formatos de texto libre, y la segunda, a partir de bases de datos. Un ejemplo pionero de utilización de datos clínicos a nivel institucional fue la plataforma MedLEE10, un sistema automatizado que permitió la obtención de información relevante a partir de los expedientes clínicos.
Otros ámbitos de enorme importancia que resultan potenciados por los instrumentos informáticos son la formación profesional y la investigación. En estos terrenos existe una enorme cantidad de fuentes de información, difíciles de manejar y seleccionar por el profesional médico. De hecho, existe una gran cantidad de publicaciones tanto en papel como electrónicas que intentan recopilar lo más relevante que se produce en el mundo editorial sobre determinada área del conocimiento médico. Un ejemplo serían las series de UpToDate11, entre las que se hallan algunas dedicadas a la medicina respiratoria (Pulmonary, Critical Care and Sleep Medicine y Allergy and Immunology, respectivamente). Algo similar ocurre con los sistemas de alerta, que se reciben por correo electrónico atendiendo a perfiles de usuario predeterminados. También en estos casos existen instrumentos de la minería de textos que permiten ir más allá en la búsqueda, la selección y el procesamiento de información.
Biología y medicina de sistemasLa «biología de sistemas» se ha definido como la ciencia dedicada al estudio sistemático de las interacciones que se producen en los sistemas biológicos. En caso de referirse al caso concreto de las enfermedades humanas se hablaría de «medicina de sistemas», aunque esta última expresión tiene también otras acepciones. Para avanzar en la biología-medicina de sistemas es necesario conectar adecuadamente el conocimiento existente en áreas diversas de la ciencia12, con el fin de aflorar nuevas propiedades e hipótesis no evidenciables a partir de los enfoques tradicionales. Los instrumentos que facilitan esta labor interdisciplinaria son en gran medida producto del desarrollo de las tecnologías de la información, que permiten el procesamiento de grandes cantidades de datos de origen diverso, desarrollando nuevo conocimiento a partir de ellos. Así, la biología-medicina de sistemas permite por ejemplo establecer modelos matemáticos de enfermedades que integran el conocimiento estructural y fisiopatológico en sus diferentes niveles de complejidad13. Entre sus objetivos se halla no solo una mejor comprensión de los procesos nosológicos, sino la posibilidad de simulación de sistemas orgánicos, para generar nuevas aproximaciones diagnósticas y terapéuticas.
La biología-medicina de sistemas utiliza a su vez diversos instrumentos. Por un lado, el objeto principal de esta revisión, la minería de textos. Esta puede definirse como un conjunto de técnicas informáticas cuyo objetivo es detectar, extraer e interpretar, de forma automatizada (o semiautomatizada), una información digital que está básicamente en formato de texto. Por otro lado, están los ya mencionados modelos matemáticos de procesos biológicos o nosológicos. Para elaborarlos, deben utilizarse instrumentos y procedimientos capaces de procesar adecuadamente la gran cantidad de información suministrada tanto por los avances en las técnicas de análisis biológico como por la existencia de amplios estudios clínicos y/o epidemiológicos. Entre las disciplinas y técnicas biomédicas que se consideran asociadas a la biología-medicina de sistemas se incluyen la genómica (estudio de los genes), la transcriptómica (de los transcriptomas), la proteómica (de la estructura y función de las proteínas), la interactómica (de las interacciones moleculares y sus consecuencias), la metabolómica (de las señales moleculares dejadas por los procesos biológicos) y la metagenómica (conjunto de genomas que conviven en un entorno). Es decir, las conocidas popularmente como «omics» u «ómicas». En el campo de la clínica y la epidemiología tenemos diversos ejemplos relacionados con el aparato respiratorio. Unos de los más recientes son los publicados por García-Aymerich et al.14 y Burgel et al.15 sobre la tipificación de fenotipos en la EPOC, a partir de un análisis de cluster sobre datos de todo tipo procedentes de pacientes reales. Una tercera faceta de la biología-medicina de sistemas, también relacionada con las ciencias «ómicas», es el análisis de los resultados generados por técnicas como los microarrays o chips de ADN y de proteínas, que se utilizan para analizar simultánea y respectivamente la expresión diferencial de gran número de estos. Son técnicas que han sido ya abundantemente utilizadas en la investigación sobre enfermedades respiratorias. Así, Steiling et al.16 y Pierrou et al.17 han demostrado los efectos del tabaco sobre la expresión de múltiples genes relacionados con la lesión celular y el estrés oxidativo en el epitelio bronquial. Sofisticando aún más la complejidad, algunos trabajos reúnen en un mismo análisis datos genéticos con datos clínicos y epidemiológicos18.
Minería de textosComo ya se ha mencionado, la minería de textos o datos es un conjunto de técnicas informáticas que permiten el procesamiento de información digital de forma automatizada. Esto permite no solo disponer de dicha información en formato manejable, sino generar nuevo conocimiento. En otros campos del saber biomédico, como la biología molecular o la propia biología de sistemas, este instrumento se ha estado utilizando desde hace años, dando lugar a interesantes resultados9,19. En su expresión más sencilla, todos realizamos minería de textos cuando empleamos una herramienta tipo PubMed20 sobre una base bibliográfica como MEDLINE, seleccionando un tema a partir de palabras clave o autores determinados21.
Una vez obtenida la información relevante a través de técnicas de minería de textos, se procede al llamado «curado». Esta es una fase que puede realizarse de forma automática, semiautomática o «manual». En el curado automático se pueden añadir requerimientos adicionales, tanto binarios (se escoge o se descarta una información de acuerdo con reglas preestablecidas) como categóricos (p.ej., dando un peso específico al factor de impacto de la revista, al tipo de artículo, al diseño o nivel de evidencia del estudio, al origen de los datos [seres humanos, modelos animales, cultivos celulares], etc.). En el curado manual, un experto en el tema depura la lista de datos o fuentes de información, según su criterio. Sin embargo, y como se discutirá más adelante, con excesiva frecuencia se considera como experto a cualquier profesional de las ciencias biomédicas, no necesariamente ducho en la faceta objeto de la búsqueda.
Un tercer punto importante, una vez seleccionada la información, es establecer conexiones entre datos dispersos. A este proceso se le denomina integración y es fundamental para generar nuevo conocimiento. Resulta obvio que la conexión e integración entre datos obtenidos en diversas áreas de conocimiento no suele producirse de forma espontánea con los métodos tradicionales. Por tanto, se hace necesario que existan métodos automatizados que seleccionen y pongan a disposición del profesional la información potencialmente relevante, independientemente del área del saber en que se haya generado.
A lo largo de los siguientes apartados se detallará cómo puede utilizarse la minería de textos o de datos en las diferentes facetas de la medicina. Entre otras, el seguimiento de hallazgos relevantes en la investigación más básica para su traslado a la clínica, el diagnóstico y la clasificación de la gravedad, y el establecimiento de un pronóstico en algunas de las entidades respiratorias más prevalentes.
Contextualización históricaAunque la necesidad de procesar gran cantidad de información de forma automática es relativamente nueva, la tecnología en que se basa la actual minería de textos tiene ya cierto recorrido. Así, hace ya medio siglo se utilizaban técnicas similares para el procesado de discursos22 o en estudios sobre estructuras lingüísticas23. En medicina, algunos de los trabajos pioneros fueron llevados a cabo por Swanson24,25, que estableció relaciones entre fenómenos aparentemente inconexos, como la migraña y el déficit de magnesio, a partir de los títulos de artículos obtenidos en la base de datos MEDLINE25. Estas relaciones fueron validadas experimentalmente años más tarde26. Con una metodología parecida, Srinivasan y Libbus27 y Weeber et al.28 determinaron el potencial efecto beneficioso de la curcumina y la talidomida en pacientes con enfermedad de Crohn. En la actualidad este tipo de búsquedas se está utilizando ampliamente en medicina y en biología9 para establecer asociaciones entre, por ejemplo, genes (o proteínas) con enfermedades29-32, o para valorar interacciones entre diversas proteínas33,34. Un instrumento interesante para este objetivo es DisGeNET, diseñado para relacionar la información procedente de las diversas «omics» con la generada sobre las distintas enfermedades31,32. En cuanto a la patología respiratoria, y como se verá más adelante, estas técnicas e instrumentos se están utilizando intensivamente para el estudio de diversos aspectos de la EPOC, el asma bronquial y el cáncer de pulmón.
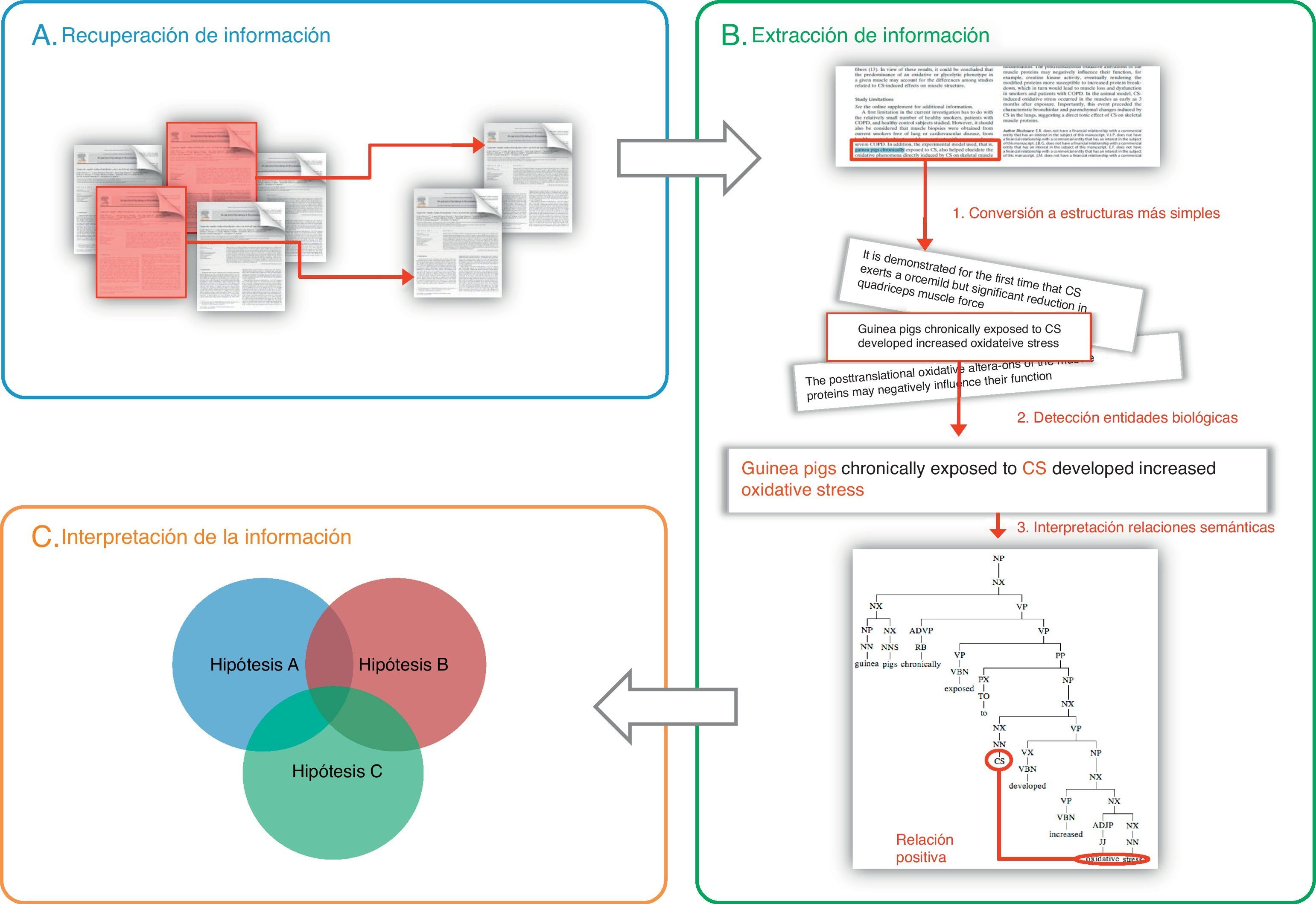
Aspectos generales de la minería de textosPor definición, la minería de textos y de datos tiene como objetivo recuperar, extraer e interpretar la información almacenada bajo formato electrónico en grandes archivos documentales y bases de datos, mediante el uso de métodos automáticos o semiautomáticos (fig. 1)9.
Esquema general de los métodos usados en la minería de textos y de datos: A)Recuperación de la información; B)Extracción de la información, y C)Interpretación de la información (en ocasiones, la integración de diversas hipótesis previamente contrastadas puede dar lugar a una nueva hipótesis conjunta). En el panel B se ejemplifican los 3pasos necesarios para extraer la información: 1)descomposición de la información en unidades básicas (p.ej., frases); 2)identificación de las entidades biológicas, y 3)interpretación de las relaciones entre las entidades biológicas.
La recuperación de la información consiste en identificar documentos que contengan datos sobre la pregunta generada. Por ejemplo, ¿cuáles son los biomarcadores que han sido involucrados o presentan potencial para el diagnóstico de la EPOC? En la actualidad, la localización de la información es un problema menor, pues existen infinidad de fuentes accesibles en formato electrónico, tanto originales (revistas, libros de texto, páginas web) como ya recopiladas en bases de datos. Un ejemplo de estas últimas es la ya mencionada MEDLINE, sobre la que actúa el sistema de recuperación de información PubMed20. Existen sistemas más específicos, como Textpresso35,36 o HLungDB37,38, que es una base de datos sobre cáncer de pulmón que agrupa información sobre genes y proteínas implicados.
El objetivo del segundo paso, la extracción de información, consiste en identificar la parte relevante de esta entre todos los datos recuperados. Esencialmente requiere 3etapas (fig. 1B): a)procesamiento, con conversión de textos complejos en estructuras simples (p.ej., palabras o frases cortas), interpretables por los sistemas informáticos; b)identificación cierta (estandarizada) de las entidades clínicas o los procesos biológicos a que hacen referencia los documentos, y c)interpretación de las relaciones semánticas entre diversas estructuras o entidades. En la extracción de información se utilizan habitualmente sistemas basados en algoritmos de inteligencia artificial39, métodos estadísticos40 o sistemas mixtos (p.ej., GENIA Tagger41,42 o Standford Log-Linear Part-of-Speech Tagger)43,44.
Un problema inherente a las fases de identificación e interpretación es que los mecanismos automáticos, que permiten filtrar el conocimiento disponible, no poseen el criterio discrecional suficiente. A esto se añade que, en general, los equipos profesionales que diseñan dichos instrumentos carecen de expertos en la materia concreta de la búsqueda. La disociación entre estos últimos y los profesionales de la bioinformática es más acusada en el caso de la medicina que en el de la biología, y ha sido identificada como el problema principal en el diseño de los instrumentos de filtrado y curado de la información. Esto es inherente a la parcelación actual de la ciencia en disciplinas, pero se agrava con la progresiva segmentación del conocimiento dentro de cada una. Es pues fundamental incorporar a profesionales clínicos en los equipos multidisciplinares de trabajo45.
Un problema adicional es que los resultados de los estudios no son axiomas, pero a menudo son tratados como tales por los profesionales procedentes de ciencias más exactas que la medicina. También es importante no olvidar que aunque la minería de datos no es una tecnología totalmente nueva y sus resultados son prometedores, aún existen aspectos que pueden mejorarse. Por ejemplo, a nivel tecnológico existe un problema inherente al lenguaje biomédico: la variabilidad léxica y semántica existente entre las diferentes disciplinas. Es un problema que aunque se puede abordar con cierto éxito mediante algoritmos de inteligencia artificial o métodos estadísticos, todavía no está completamente resuelto. Otro aspecto a tener en cuenta, principalmente en el uso de la minería de textos en medicina, es que las conclusiones obtenidas pueden acabar teniendo efecto sobre los pacientes, con lo que es importante que estén completamente contrastadas.
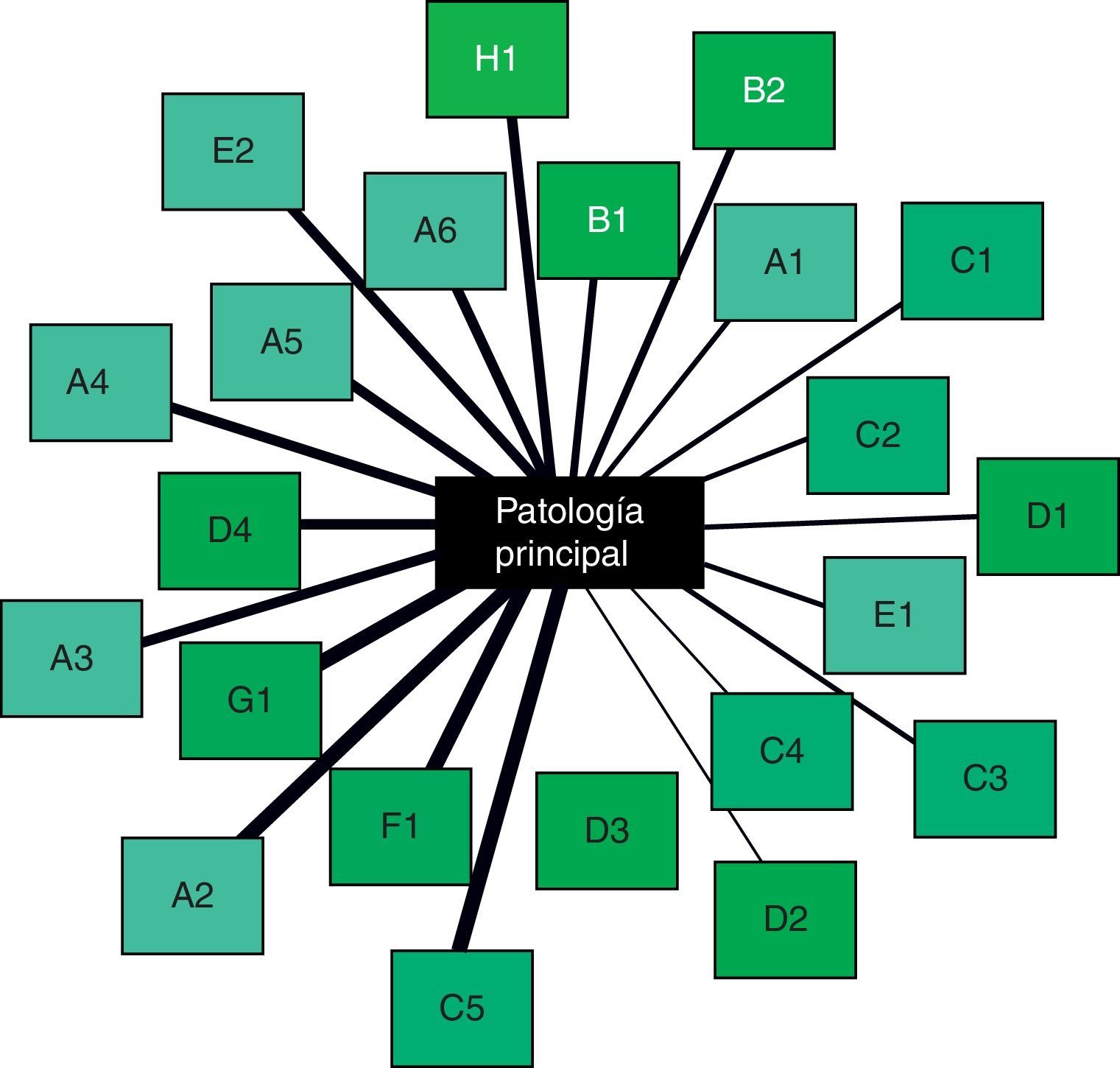
El último paso es el de la de interpretación de resultados. Con él se persigue integrar la información obtenida en los pasos anteriores, y en última instancia obtener asociaciones entre entidades o fenómenos biológicos o clínicos, inicialmente no detectables mediante los métodos convencionales. Volviendo al estudio de Swanson25, la integración de diversas hipótesis contrastadas individualmente es lo que permitió originar una nueva hipótesis que vinculaba el déficit de magnesio con la migraña. En la figura 2 se muestra como ejemplo una representación gráfica de las asociaciones entre una entidad (la EPOC) y sus comorbilidades. Este tipo de imágenes facilita la interpretación simultánea de información diversa obtenida con la minería de datos, generando nuevas hipótesis.
Representación gráfica de las relaciones entre una entidad principal (centro) y diversas comorbilidades potenciales (recuadros de la periferia). El grosor de las líneas entre uno y otros expresa la cantidad de información disponible en cada caso, tras el curado de los datos seleccionados automáticamente. Estas relevancias relativas se han ordenado según rotación horaria. Las comorbilidades de un mismo sistema o aparato del organismo se muestran con un patrón común en el relleno de los recuadros y como letra correlativa de identificación.
Como ya se ha comentado, la alta producción y disponibilidad de datos en el ámbito de la medicina y de las ciencias biológicas más básicas hace de él un espacio apropiado para el uso de la minería de textos. En el campo concreto de las enfermedades respiratorias han aparecido en los últimos años numerosos estudios que hacen uso de esta técnica y que abarcan las diversas etapas que van desde el conocimiento más básico a la medicina aplicada.
Conocimiento en ciencias básicasEn campos como la biología molecular o la fisiología, la minería de textos está originando una transición del modelo científico deductivo clásico —en el que una hipótesis se verifica experimentalmente— a un modelo basado en la búsqueda «a ciegas» de asociaciones entre hechos aparentemente no conectados, pero validables experimentalmente46,47. Para ello es conveniente el desarrollo de plataformas especializadas. Así, existen bases de datos unificadas, como la ya mencionada HLungDB37,38, que han integrado información sobre genes, proteínas, modificaciones epigenéticas y características clínicas, relacionada en todos los casos con el cáncer de pulmón. El principal objetivo de esta plataforma es establecer una red de conexiones basada en moléculas asociadas con esta entidad, facilitando así tanto la investigación más básica como la integración con áreas relativamente distantes como la clínica o la epidemiología. Esto resulta fundamental en entidades como el cáncer de pulmón, que implican alteraciones complejas y de causa multifactorial. Pueden así desarrollarse vías de estudio previamente inexploradas o sugerirse nuevas alternativas terapéuticas. También hay numerosos ejemplos del uso de la minería de textos en aspectos básicos relacionados con la EPOC. Así, Comandini et al.48 utilizaron datos correspondientes a genes y a expresión de proteínas en sujetos no fumadores, fumadores sin enfermedad pulmonar y fumadores con EPOC, con el objetivo de identificar efectos inducidos por el tabaco. Sus resultados sugieren que determinados genes con actividad antioxidante se sobreexpresan ante la exposición al tabaco en algunos individuos y podrían jugar un papel importante en la protección tisular frente al estrés oxidativo. Estos genes o sus productos podrían ser utilizados como biomarcadores negativos del riesgo para desarrollar una EPOC. En un trabajo muy reciente de algunos miembros de nuestro grupo49 se ha utilizado la minería de textos para estudiar las comorbilidades de esta misma enfermedad y los mecanismos potencialmente implicados, aflorando la sorprendente abundancia de datos en algunas de las asociaciones (como la de EPOC con cardiopatía isquémica o con cáncer de pulmón) y la escasa presencia en la literatura de otras (como es el caso de las de EPOC con alteraciones nutricionales o con hipertensión pulmonar). También en el caso del asma bronquial se han publicado trabajos interesantes. Por ejemplo, Su et al.50 han investigado las relaciones de diversos genes entre sí, y de estos con el ambiente en el asma infantil, concluyendo que algunos de ellos condicionan la susceptibilidad a desarrollar asma en los niños. Tremblay et al.51, por su parte, implementaron una metodología (Genes to Diseases [G2D]) basada en la minería de textos para identificar genes candidatos a estar implicados en el desarrollo de asma y atopia. En cuanto a la neumonía y al síndrome del distrés respiratorio del adulto (SDRA) sucede algo parecido. Frenzel et al.52, analizando múltiples marcadores en el lavado broncoalveolar de pacientes con SDRA, demostraron que la determinación de IL-6 puede ser de gran valor pronóstico.
Parece claro que el análisis automático del conocimiento procedente de diversas ciencias básicas puede aportar nueva luz sobre las enfermedades respiratorias.
Diagnóstico y manejo clínico del paciente respiratorioEstablecer un diagnóstico de certeza y categorizar la gravedad es crucial en cualquier enfermedad. No obstante, esta no es siempre una tarea fácil, sobre todo si el diagnóstico requiere técnicas complejas o que precisan un entrenamiento especial. Este es el caso de la EPOC, donde se necesita tanto la colaboración del paciente como una adecuada maniobra de espirometría forzada. En el momento actual, en algunas iniciativas, como la Global Initiative for Chronic Obstructive Lung Disease (GOLD)53, se requiere además conocer el número de ingresos recientes y la cuantía de la disnea. Utilizar un análisis automático de datos puede permitir verificar la bondad de una determinada maniobra espirométrica, a la vez que aproximar el diagnóstico completo basado en otras variables. Matsumoto et al.54, mediante minería de datos efectuada en registros electrónicos de unos 27.000 pacientes, observaron que la obstrucción al flujo aéreo detectable por los valores espirométricos no había sido diagnosticada en hasta el 86% de los casos. Este es un ejemplo de algunas aplicaciones de la minería de datos, que permiten además establecer alarmas diagnósticas en las historias clínicas, evitando que hallazgos relevantes pasen desapercibidos al profesional asistencial. Por otra parte, y como se ha visto, el análisis automático de datos puede permitir hallar nuevas relaciones entre variables, generando hipótesis que conduzcan a sistemas alternativos o complementarios de diagnóstico. Respecto del cáncer de pulmón, existen diversos pasos cruciales en su detección y posterior estratificación. Uno de ellos es la valoración de la afectación ganglionar, que se realiza inicialmente mediante técnicas de imagen (tomografía axial computarizada [TAC] y tomografía por emisión de positrones), y se confirma o descarta mediante la obtención de muestras tisulares por ultrasonografía endobronquial o mediastinoscopia. Lu et al.55 proponen un nuevo método automatizado para la orientación inicial de la afectación ganglionar. Este se basa también en la TAC, pero utilizando luego la minería de datos para comparar los hallazgos con los registros anatómicos de nódulos linfáticos pertenecientes a pacientes ya diagnosticados.
Una fase del proceso diagnóstico que es complementaria a la identificación de la enfermedad es el establecimiento de un pronóstico. Esto implica generalmente la categorización de la gravedad o de la extensión de la enfermedad. En esta fase es igualmente importante disponer de buenos métodos de asignación a cada categoría, pues con frecuencia condicionará el tratamiento. En los últimos años han aparecido sistemas de clasificación automática de las etapas del cáncer de pulmón, que utilizan datos clínicos e histológicos registrados en informes diversos de una o varias instituciones56,57. En el caso del trasplante de pulmón o combinado de corazón-pulmón, una adecuada predicción de la supervivencia es crítica no solo para un paciente concreto, sino también para la selección de donantes y receptores. Existe en la actualidad una gran cantidad de información referente a trasplantes (procedimientos, monitorización de pacientes, etc.) que se ha utilizado junto con métodos estadísticos clásicos para realizar predicciones de morbilidad y supervivencia respecto de diversos órganos, incluido el pulmón58. Más recientemente se está utilizando también la minería de datos59, ya que presenta ciertas ventajas sobre los métodos estadísticos clásicos. Por ejemplo, no está limitada por el número de observaciones ni requiere independencia de los observadores, como sucedería en un estudio concreto.
Un campo en el que la minería de datos ha sido particularmente fecunda es el del enfermo respiratorio semicrítico y crítico. >A lo largo de las últimas décadas las unidades a cargo de estos pacientes han implementado sus sistemas de recogida de datos, y como consecuencia directa se han desarrollado 2aplicaciones fundamentales basadas en la minería de textos.
Por un lado, la generación de modelos predictivos a corto y medio plazo, que permiten crear alertas inteligentes y sistemas de toma de decisiones. Entre los numerosos estudios dedicados al paciente crítico destacan el de Tzavaras et al.60, que mediante minería de datos y redes neuronales desarrollaron un modelo de soporte a la decisión clínica, basado en la identificación de las variables fisiológicas clave para decidir la instauración de ventilación mecánica.
Por otra parte, se han desarrollado también modelos de largo plazo, utilizados principalmente como método de evaluación de la calidad de los equipos e instituciones implicadas en el cuidado del paciente respiratorio crítico61-63. Algunas de estas aplicaciones se han extendido posteriormente a unidades de hospitalización convencional, como las salas de neumología y de cirugía torácica. Tradicionalmente, estos instrumentos se han construido utilizando únicamente datos demográficos y administrativos, para determinar índices de supervivencia, de mortalidad, etc. Sin embargo, de forma más reciente, se están aprovechando los datos fisiológicos y clínicos que quedan almacenados para generar modelos basados en la minería de datos. Existen diversos estudios, como los de Bohensky et al.62 o Kim et al.63, que han demostrado que estos modelos son superiores a los métodos clásicos en la predicción de supervivencia.
Las vías clínicas son el registro de los procedimientos realizados sobre un paciente concreto durante su estancia hospitalaria. También en este campo se han publicado estudios donde se emplean métodos de análisis automatizado de datos64,65. Las vías clínicas constituyen una herramienta básica de gestión de la calidad asistencial y se ha demostrado que su implementación permite disminuir la variabilidad en la práctica clínica. Otras ventajas son la ayuda al clínico en la toma de decisiones y una optimización de los recursos empleados, con reducción del tiempo y costes en la atención.
ConclusionesSe dispone hoy en día de gran cantidad de información científica y médica. No obstante, existe un problema inherente a tal volumen de datos: su recuperación selectiva e interpretación se hacen prácticamente imposibles para un profesional si emplea los métodos clásicos. En ese escenario, el uso de herramientas bioinformáticas como la minería de textos o datos adquiere una relevancia fundamental. Estas herramientas, que han tenido ya un papel importante en otros campos del saber biomédico, se han empezado a utilizar recientemente en la medicina respiratoria. Los niveles más evidentes de aplicación médica (en investigación y en clínica) de la minería de textos son la integración y la transferencia de los avances obtenidos en las ciencias más básicas, y una mejor comprensión de los procesos de diagnóstico, de categorización de la gravedad y de establecimiento de pronóstico de las enfermedades. También puede ser de gran utilidad para generar modelos predictivos de outcomes, crear alertas inteligentes y ayudar al clínico en la toma de decisiones.
FinanciaciónCB06/06/0043 (CIBERES), SAF2011-26908 (Ministerio de Economía y Competitividad) y 2009SGR393 (Generalitat de Catalunya).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Deseamos agradecer al Dr. Ferran Sanz y la Dra. Laura Furlong, del Programa de Investigación en Informática (GRIB, IMIM-UPF), por su asesoramiento y consejos en la redacción del presente manuscrito.